Visualização de dados com seaborn¶
No capítulo Plotagem e formatação de gráficos, fizemos uma breve introdução à visualização de dados e aprendemos a utilizar o matplotlib para realizar plotagens básicas de gráficos. Neste capítulo, exploraremos a visualização de dados utilizando o módulo seaborn.
O seaborn estende as potencialidades do matplotlib em, praticamente, dois aspectos: i) provendo melhor estilização dos gráficos e tornando-os visualmente “belos”; ii) compactando funções de plotagem do matplotlib, de modo que plotagens robustas sejam obtidas com menos instruções de código.
Vamos começar importando as bibliotecas que utilizaremos.
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
import warnings
warnings.filterwarnings('ignore')
Vamos reconstruir e importar alguns DataFrames.
serie_Idade = pd.Series({'Ana':20, 'João': 19, 'Maria': 21, 'Pedro': 22, 'Túlio': 20}, name="Idade")
serie_Peso = pd.Series({'Ana':55, 'João': 80, 'Maria': 62, 'Pedro': 67, 'Túlio': 73}, name="Peso")
serie_Altura = pd.Series({'Ana':162, 'João': 178, 'Maria': 162, 'Pedro': 165, 'Túlio': 171}, name="Altura")
dicionario_series_exemplo = {'Idade': serie_Idade, 'Peso': serie_Peso, 'Altura': serie_Altura}
df_dict_series = pd.DataFrame(dicionario_series_exemplo);df_dict_series
| Idade | Peso | Altura | |
|---|---|---|---|
| Ana | 20 | 55 | 162 |
| João | 19 | 80 | 178 |
| Maria | 21 | 62 | 162 |
| Pedro | 22 | 67 | 165 |
| Túlio | 20 | 73 | 171 |
df_exemplo = pd.read_csv('../database/exemplo_data.csv', index_col=0)
df_exemplo['coluna_3'] = pd.Series([1,2,3,4,5,6,7,8,np.nan,np.nan],index=df_exemplo.index)
df_exemplo.index = pd.to_datetime(df_exemplo.index)
df_exemplo
| coluna_1 | coluna_2 | coluna_3 | |
|---|---|---|---|
| 2020-01-01 | -0.416092 | 1.810364 | 1.0 |
| 2020-01-02 | -0.137970 | 2.578520 | 2.0 |
| 2020-01-03 | 0.575827 | 0.060866 | 3.0 |
| 2020-01-04 | -0.017367 | 1.299587 | 4.0 |
| 2020-01-05 | 1.384279 | -0.381732 | 5.0 |
| 2020-01-06 | 0.549706 | -1.308789 | 6.0 |
| 2020-01-07 | -0.282296 | -1.688979 | 7.0 |
| 2020-01-08 | -0.989730 | -0.028121 | 8.0 |
| 2020-01-09 | 0.275582 | -0.177659 | NaN |
| 2020-01-10 | 0.685132 | 0.502535 | NaN |
covid_PB = pd.read_csv('https://superset.plataformatarget.com.br/superset/explore_json/?form_data=%7B%22slice_id%22%3A1550%7D&csv=true',
sep=',', index_col=0)
covid_PB.head()
| casosAcumulados | casosNovos | descartados | recuperados | obitosAcumulados | obitosNovos | Letalidade | |
|---|---|---|---|---|---|---|---|
| data | |||||||
| 2021-10-26 | 445012 | 50 | 484906 | 337941 | 9399 | 1.0 | 0.0211 |
| 2021-10-25 | 444962 | 33 | 484859 | 337928 | 9398 | 3.0 | 0.0211 |
| 2021-10-24 | 444929 | 177 | 484859 | 337925 | 9395 | 4.0 | 0.0211 |
| 2021-10-23 | 444752 | 184 | 484853 | 337695 | 9391 | 5.0 | 0.0211 |
| 2021-10-22 | 444568 | 216 | 484830 | 337646 | 9386 | 1.0 | 0.0211 |
covid_BR = pd.read_excel('../database/HIST_PAINEL_COVIDBR_25jul2020.xlsx')
covid_BR.head()
| Unnamed: 0 | regiao | estado | municipio | coduf | codmun | codRegiaoSaude | nomeRegiaoSaude | data | semanaEpi | populacaoTCU2019 | casosAcumulado | casosNovos | obitosAcumulado | obitosNovos | Recuperadosnovos | emAcompanhamentoNovos | interior/metropolitana | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-25 | 9 | 210147125 | 0 | 0 | 0 | 0 | NaN | NaN | NaN |
| 1 | 1 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-26 | 9 | 210147125 | 1 | 1 | 0 | 0 | NaN | NaN | NaN |
| 2 | 2 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-27 | 9 | 210147125 | 1 | 0 | 0 | 0 | NaN | NaN | NaN |
| 3 | 3 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-28 | 9 | 210147125 | 1 | 0 | 0 | 0 | NaN | NaN | NaN |
| 4 | 4 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-29 | 9 | 210147125 | 2 | 1 | 0 | 0 | NaN | NaN | NaN |
Gráficos de Linha e de Dispersão¶
Através da função relplot, podemos alterar o valor do parâmetro kind para
obter gráficos do linha, com kind = 'line', ou gráficos de dispersão, com kind = 'scatter'. De modo alternativo, poderíamos também utilizar as funções lineplot e scatterplot. Entretanto, com relplot, diversos elementos de construção de figura existentes no matplotlib já são pré-configurados no seaborn. Isto é chamado no seaborn de figure-level plot.
Inicialmente, vejamos como a função lineplot se comporta como qualquer outra do matplotlib.pyplot.
fig, ax = plt.subplots()
ax = sns.lineplot(x="index", y="coluna_1", data=df_exemplo.reset_index(), label = 'Coluna 1')
ax = sns.lineplot(x="index", y="coluna_2", data=df_exemplo.reset_index(), label = 'Coluna 2')
ax = sns.lineplot(x="index", y="coluna_3", data=df_exemplo.reset_index(), label = 'Coluna 3')
ax.set_xlabel('Data')
ax.set_ylabel('Valor')
fig.autofmt_xdate() # auto-formata xticks tipo "data"
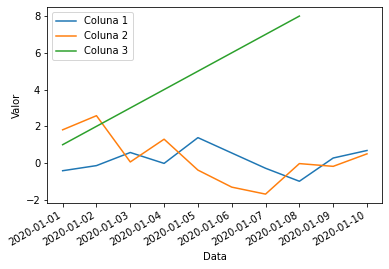
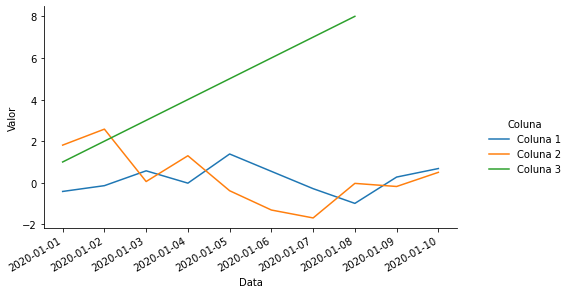
Para utilizar a função relplot, precisaremos reorganizar o banco de dados de modo que haja apenas uma coluna de valores no eixo y (Valor) e uma coluna com um identificador de cor/tonalidade (Coluna).
# reorganiza para 'Coluna 1'
df_exemplo_px = pd.DataFrame(df_exemplo['coluna_1']).rename({'coluna_1':'Valor'}, axis=1)
df_exemplo_px['Coluna'] = 'Coluna 1'
# concatena 'Coluna 2'
df_exemplo_px_temp = pd.DataFrame(df_exemplo['coluna_2']).rename({'coluna_2':'Valor'}, axis=1)
df_exemplo_px_temp['Coluna'] = 'Coluna 2'
df_exemplo_px = pd.concat([df_exemplo_px, df_exemplo_px_temp])
# concatena 'Coluna 3'
df_exemplo_px_temp = pd.DataFrame(df_exemplo['coluna_3']).rename({'coluna_3':'Valor'}, axis=1)
df_exemplo_px_temp['Coluna'] = 'Coluna 3'
df_exemplo_px = pd.concat([df_exemplo_px, df_exemplo_px_temp])
df_exemplo_px.head()
| Valor | Coluna | |
|---|---|---|
| 2020-01-01 | -0.416092 | Coluna 1 |
| 2020-01-02 | -0.137970 | Coluna 1 |
| 2020-01-03 | 0.575827 | Coluna 1 |
| 2020-01-04 | -0.017367 | Coluna 1 |
| 2020-01-05 | 1.384279 | Coluna 1 |
# 'hue' atribui as tonalidades de cor com base no identificador
p = sns.relplot(x = 'index', y='Valor', hue = 'Coluna', data=df_exemplo_px.reset_index().dropna(), kind='line')
p.fig.autofmt_xdate()
p.ax.set_xlabel('Data')
p.fig.set_size_inches(8,4) # largura, altura
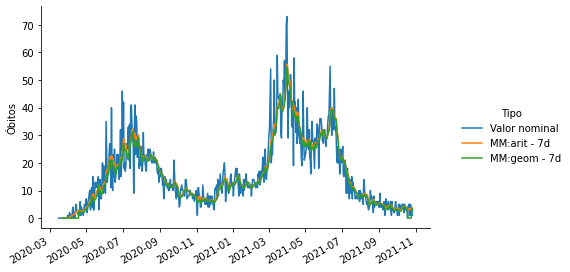
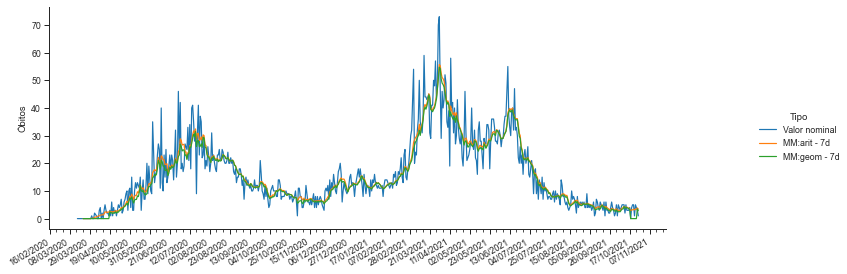
Vamos agora plotar o gráfico de óbitos por COVID-19 na Paraíba juntamente com a média aritmética móvel de 7 dias e com a média geométrica móvel de 7 dias.
Utilizaremos o método rolling de uma Series ou DataFrame do pandas. Este método cria janelas móveis onde podemos aplicar uma função agregadora (tal como média ou média geométrica).
Comentários:
A média aritmética tem a desvantagem de linearizar o efeito do crescimento ou decrescimento do número de óbitos, onde sabemos que o efeito é exponencial.
A média geométrica móvel tem a desvantagem de se anular se o número de óbitos em algum dos dias da janela for zero.
Em geral, as duas médias ficam muito próximas.
# função que calcula a média geométrica
from scipy.stats import gmean
# série
covid_PB_obitos = covid_PB.obitosNovos
covid_PB_obitos = covid_PB_obitos.sort_index()
covid_PB_obitos.name = 'Óbitos'
# dataframe
covid_PB_obitos_df = pd.DataFrame(covid_PB_obitos)
covid_PB_obitos_df['Tipo'] = 'Valor nominal'
# média aritmética móvel
covid_PB_obitos_df_temp = pd.DataFrame(covid_PB_obitos.rolling(7).mean().dropna())
covid_PB_obitos_df_temp['Tipo'] = 'MM:arit - 7d'
covid_PB_obitos_df = pd.concat([covid_PB_obitos_df, covid_PB_obitos_df_temp])
# média geométrica móvel
covid_PB_obitos_df_temp = pd.DataFrame(covid_PB_obitos.rolling(7).aggregate(gmean).dropna())
covid_PB_obitos_df_temp['Tipo'] = 'MM:geom - 7d'
covid_PB_obitos_df = pd.concat([covid_PB_obitos_df, covid_PB_obitos_df_temp])
covid_PB_obitos_df.index = pd.to_datetime(covid_PB_obitos_df.index)
covid_PB_obitos_df.tail()
| Óbitos | Tipo | |
|---|---|---|
| data | ||
| 2021-10-22 | 0.000000 | MM:geom - 7d |
| 2021-10-23 | 0.000000 | MM:geom - 7d |
| 2021-10-24 | 0.000000 | MM:geom - 7d |
| 2021-10-25 | 3.465261 | MM:geom - 7d |
| 2021-10-26 | 2.842676 | MM:geom - 7d |
p = sns.relplot(x = 'data', y='Óbitos',
hue = 'Tipo',
data=covid_PB_obitos_df.reset_index(),
kind='line')
p.fig.autofmt_xdate()
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
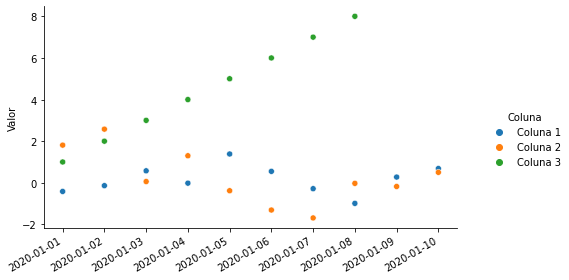
Vamos agora construir um gráfico de dispersão com o DataFrame df_exemplo_px.
# 'scatter' é padrão
p = sns.relplot(x = 'index', y='Valor',
hue = 'Coluna',
data=df_exemplo_px.reset_index().dropna())
p.fig.autofmt_xdate()
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
Vamos forçar os limites de datas a ficarem dentro do mínimo (menos um dia) e do máximo (mais um dia):
p = sns.relplot(x = 'index', y='Valor',
hue = 'Coluna',
data=df_exemplo_px.reset_index().dropna())
# offset de data
p.ax.set_xlim((df_exemplo_px.reset_index()['index'].min()
- pd.DateOffset(days=1)),
(df_exemplo_px.reset_index()['index'].max()
+ pd.DateOffset(days=1)))
p.fig.autofmt_xdate()
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
# especifica cores e tamanhos
p = sns.relplot(x = 'index', y='coluna_1',
hue = 'coluna_2',
size = 'coluna_3',
data=df_exemplo.reset_index().dropna())
p.ax.set_xlim((df_exemplo.reset_index()['index'].min()
- pd.DateOffset(days=1)),
(df_exemplo.reset_index()['index'].max()
+ pd.DateOffset(days=1)))
p.fig.autofmt_xdate()
p.ax.set_xlabel('')
p.fig.set_size_inches(8,4)
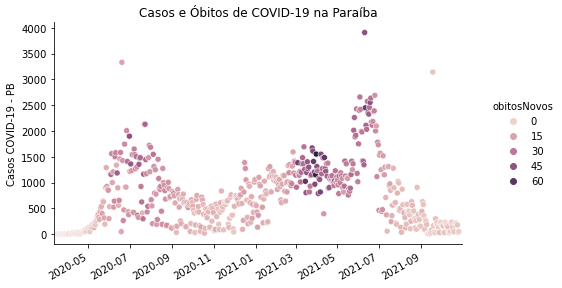
covid_PB_casos_obitos = covid_PB[['obitosNovos', 'casosNovos']].sort_index()
covid_PB_casos_obitos.index = pd.to_datetime(covid_PB_casos_obitos.index)
# cor: óbitos novos
p = sns.relplot(x = 'data', y = 'casosNovos',
hue = 'obitosNovos',
data=covid_PB_casos_obitos.reset_index())
p.ax.set_xlim((covid_PB_casos_obitos.reset_index()['data'].min()
- pd.DateOffset(days=5)),
(covid_PB_casos_obitos.reset_index()['data'].max()
+ pd.DateOffset(days=5)))
p.fig.autofmt_xdate()
p.ax.set_xlabel('')
p.ax.set_ylabel('Casos COVID-19 - PB')
p.ax.set_title('Casos e Óbitos de COVID-19 na Paraíba')
p.fig.set_size_inches(8,4)
Gráficos de dispersão em dados categóricos¶
Quando há muitos valores repetidos em uma variável, os gráficos de dispersão podem não ilustrar efetivamente o comportamento dos dados. Neste caso, é interessante que o gráfico considere a repetição dos valores dentro de uma mesma categoria.
Comentários:
Isto acontece quando o eixo horizontal contém variáveis categóricas e, assim, tem-se repetição de valores dentro de uma mesma categoria.
Para gráficos deste tipo, utilizaremos os dados de óbitos por COVID-19 no Brasil. Agruparemos o número de óbitos por dia da semana.
covid_BR_obitos = covid_BR.query('regiao == "Brasil"')[['obitosNovos','data']]
covid_BR_obitos.data = pd.to_datetime(covid_BR_obitos.data)
covid_BR_obitos['Dia'] = covid_BR_obitos.data.dt.weekday.map(
{0:'Segunda-Feira',
1:'Terça-Feira',
2:'Quarta-Feira',
3:'Quinta-Feira',
4:'Sexta-Feira',
5:'Sábado',
6:'Domingo'})
covid_BR_obitos = covid_BR_obitos.set_index('data')
covid_BR_obitos
| obitosNovos | Dia | |
|---|---|---|
| data | ||
| 2020-02-25 | 0 | Terça-Feira |
| 2020-02-26 | 0 | Quarta-Feira |
| 2020-02-27 | 0 | Quinta-Feira |
| 2020-02-28 | 0 | Sexta-Feira |
| 2020-02-29 | 0 | Sábado |
| ... | ... | ... |
| 2020-07-21 | 1367 | Terça-Feira |
| 2020-07-22 | 1284 | Quarta-Feira |
| 2020-07-23 | 1311 | Quinta-Feira |
| 2020-07-24 | 1156 | Sexta-Feira |
| 2020-07-25 | 1211 | Sábado |
152 rows × 2 columns
Se quisermos determinar a ordem do eixo x, relplot não é a função ideal. Além disso, devido à sobreposição dos dados, ela definitivamente não é a ideal para variáveis categóricas. Vejamos:
p = sns.relplot(x='Dia', y='obitosNovos', data=covid_BR_obitos)
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
O gráfico stripplot¶
O stripplot é um gráfico de dispersão onde a cada observação é colocado um deslocamento aleatório para evitar a sobreposição e fornecer uma ideia mais precisa da quantidade de dados.
Vamos construir o stripplot através da função catplot. O stripplot é o gráfico padrão do catplot (tem o argumento kind = 'strip').
Podemos determinar a ordem das variáveis categóricas com o argumento order.
p = sns.catplot(x='Dia', y='obitosNovos',
data=covid_BR_obitos, order =
['Segunda-Feira', 'Terça-Feira',
'Quarta-Feira', 'Quinta-Feira',
'Sexta-Feira', 'Sábado', 'Domingo'])
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
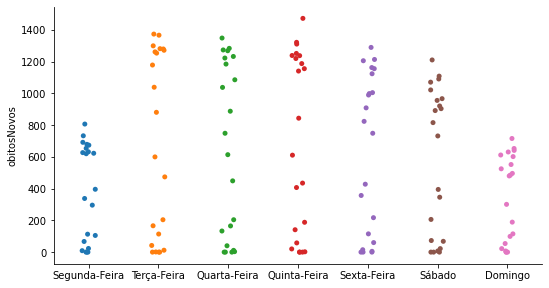
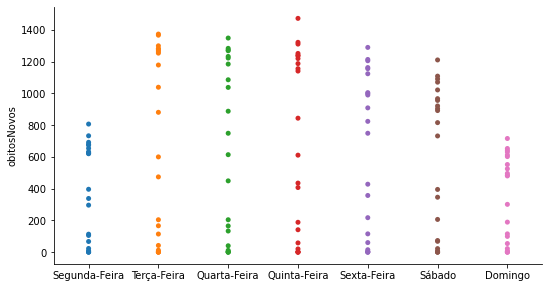
Se colocarmos jitter=False, obteremos o gráfico de dispersão usual (com o detalhe de que podemos definir a ordem dos rótulos do eixo x).
p = sns.catplot(x='Dia', y='obitosNovos',
jitter = False,
data=covid_BR_obitos,
order = ['Segunda-Feira', 'Terça-Feira',
'Quarta-Feira', 'Quinta-Feira',
'Sexta-Feira', 'Sábado', 'Domingo'])
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
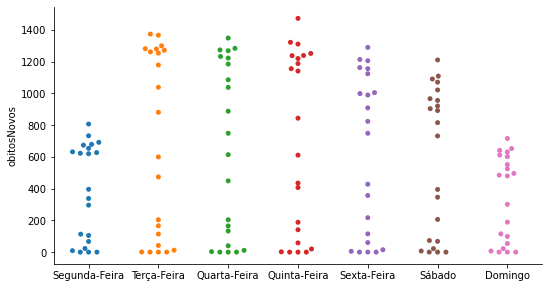
O gráfico swarmplot¶
O swarmplot é um gráfico de dispersão onde, diferentemente do stripplot, nenhum dado pode ficar sobreposto. Desta forma, também fornece uma ideia mais precisa da quantidade de dados.
Construiremos o swarmplot através da função catplot com o argumento kind = 'swarm'. Como o swarmplot também é um tipo do catplot, podemos determinar a ordem das variáveis categóricas com o argumento order.
p = sns.catplot(x='Dia', y='obitosNovos',
kind = 'swarm',
data=covid_BR_obitos,
order = ['Segunda-Feira', 'Terça-Feira',
'Quarta-Feira', 'Quinta-Feira',
'Sexta-Feira', 'Sábado', 'Domingo'])
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
Gráficos de Barras e Colunas¶
Para criar gráficos de barras e colunas com o seaborn utilizaremos a função catplot com o argumento kind=bar. Se a variável categórica estiver no eixo x, o gráfico será de coluna; se a variável categórica estiver no eixo y, o gráfico será de barra.
covid_Regioes = covid_BR[['regiao','obitosNovos']].groupby('regiao').sum().query('regiao != "Brasil"')/2
p = sns.catplot(x='regiao', y='obitosNovos',
kind = 'bar',data=covid_Regioes.reset_index())
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
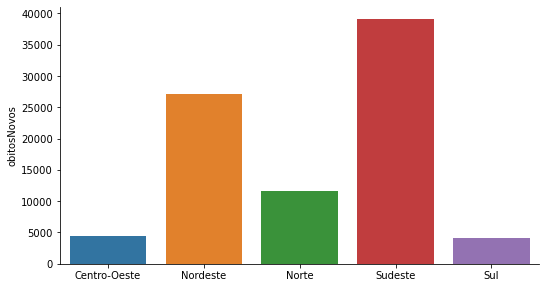
covid_Regioes = covid_BR[['regiao','obitosNovos']].groupby('regiao').sum().query('regiao != "Brasil"')/2
p = sns.catplot(x='obitosNovos', y='regiao',
kind = 'bar',data=covid_Regioes.reset_index())
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
df_dict_series_sns = pd.DataFrame(df_dict_series.Idade).rename({'Idade':'Valor'}, axis=1)
df_dict_series_sns['Dado'] = 'Idade'
df_dict_series_sns_temp = pd.DataFrame(df_dict_series.Altura).rename({'Altura':'Valor'}, axis=1)
df_dict_series_sns_temp['Dado'] = 'Altura'
df_dict_series_sns = pd.concat([df_dict_series_sns, df_dict_series_sns_temp])
df_dict_series_sns_temp = pd.DataFrame(df_dict_series.Peso).rename({'Peso':'Valor'}, axis=1)
df_dict_series_sns_temp['Dado'] = 'Peso'
df_dict_series_sns = pd.concat([df_dict_series_sns, df_dict_series_sns_temp])
df_dict_series_sns
| Valor | Dado | |
|---|---|---|
| Ana | 20 | Idade |
| João | 19 | Idade |
| Maria | 21 | Idade |
| Pedro | 22 | Idade |
| Túlio | 20 | Idade |
| Ana | 162 | Altura |
| João | 178 | Altura |
| Maria | 162 | Altura |
| Pedro | 165 | Altura |
| Túlio | 171 | Altura |
| Ana | 55 | Peso |
| João | 80 | Peso |
| Maria | 62 | Peso |
| Pedro | 67 | Peso |
| Túlio | 73 | Peso |
p = sns.catplot(x='index', y='Valor',
hue='Dado',
data = df_dict_series_sns.reset_index(),
kind='bar')
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
Box Plot e plots alternativos¶
Tanto o BoxPlot quanto os plots alternativos que apresentaremos aqui (violinplot e boxenplot) fazem parte do catplot. Para construir um
Box Plot utiliza-se o argumento
kind='box';Violin Plot utiliza-se o argumento
kind='violin';Boxen Plot (ou letter-value plot) utiliza-se o argumento
kind='boxen'.
Note
O boxenplot foi criado por Hadley Wickham (criador do ggplot2 e da maioria dos pacotes do tidyverse do R) e colaboradores e é uma generalização do BoxPlot que apresenta mais quantis. Foi introduzido como letter-value plots. O violinplot recebe este nome, pois seu gráfico assemelha-se a um violino.
p = sns.catplot(x='Dia', y='obitosNovos',
kind = 'box',
data=covid_BR_obitos,
order = ['Segunda-Feira', 'Terça-Feira',
'Quarta-Feira', 'Quinta-Feira',
'Sexta-Feira', 'Sábado', 'Domingo'])
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
covid_regioes_diarios_px = covid_BR.set_index(
'data').query('regiao != "Brasil"')[['obitosNovos', 'regiao']].reset_index().rename(
{'obitosNovos':'Óbitos','regiao':'Região','data':'Data'},axis=1)
covid_regioes_diarios_px = covid_regioes_diarios_px.groupby(['Região','Data']).sum()/2
covid_regioes_diarios_px = covid_regioes_diarios_px.reset_index().set_index('Data')
covid_regioes_diarios_px
| Região | Óbitos | |
|---|---|---|
| Data | ||
| 2020-02-25 | Centro-Oeste | 0.0 |
| 2020-02-26 | Centro-Oeste | 0.0 |
| 2020-02-27 | Centro-Oeste | 0.0 |
| 2020-02-28 | Centro-Oeste | 0.0 |
| 2020-02-29 | Centro-Oeste | 0.0 |
| ... | ... | ... |
| 2020-07-21 | Sul | 166.0 |
| 2020-07-22 | Sul | 146.0 |
| 2020-07-23 | Sul | 165.0 |
| 2020-07-24 | Sul | 123.0 |
| 2020-07-25 | Sul | 151.0 |
760 rows × 2 columns
p = sns.catplot(x='Região', y='Óbitos',
kind = 'box',
data=covid_regioes_diarios_px)
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
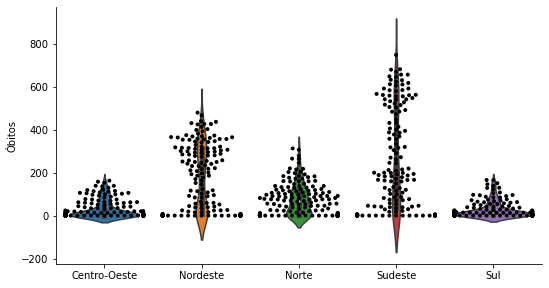
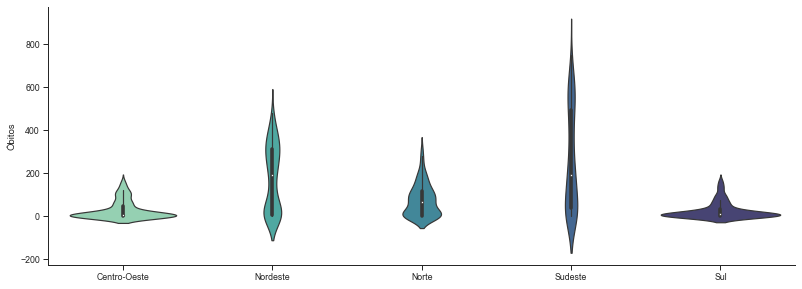
Na presença de muitos outliers, como é o caso do gráfico anterior, é interessante considerar uma alternativa ao Box Plot.
Vamos ver agora o Boxen Plot (ou letter-value plots). Este plot considera os quantis: …, 0.8%, 1.56%, 3.13%, 6.25%, 12.5%, 25%, 50%, 75%, 87.5%, 93.75%, 96.88%, 98.44%, 99.24%, …
p = sns.catplot(x='Região', y='Óbitos',
kind = 'boxen',
data=covid_regioes_diarios_px)
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)

Porém, em um gráfico sem muitos outliers, o Boxen Plot não difere muito do Box Plot.
p = sns.catplot(x='Dia', y='obitosNovos',
kind = 'boxen',
data=covid_BR_obitos,
order = ['Segunda-Feira', 'Terça-Feira',
'Quarta-Feira', 'Quinta-Feira',
'Sexta-Feira', 'Sábado', 'Domingo'])
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
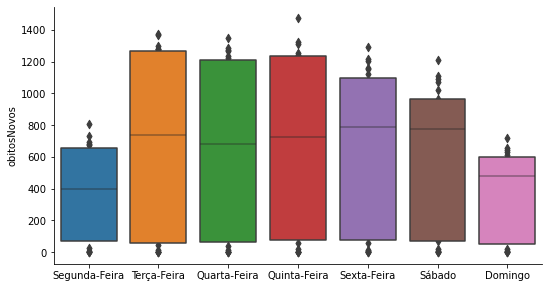
Na presença de muitos outliers, também é preferível um Violin Plot em vez de um Box Plot, para tornar visível o que está ocorrendo.
p = sns.catplot(x='Região', y='Óbitos',
kind = 'violin',
data=covid_regioes_diarios_px)
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)

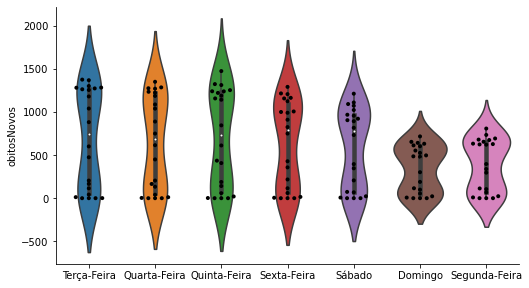
Muitas vezes, é interessante sobrepor um Violin Plot a um Swarm Plot para evidenciar o comportamento da distribuição dos dados.
p = sns.catplot(x='Região', y='Óbitos',
kind = 'violin',
data=covid_regioes_diarios_px)
sns.swarmplot(x='Região', y='Óbitos',
data=covid_regioes_diarios_px,
ax = p.ax,
size=4, color='k')
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
p = sns.catplot(x='Dia', y='obitosNovos',
kind = 'violin',
data=covid_BR_obitos)
sns.swarmplot(x='Dia', y='obitosNovos',
data=covid_BR_obitos,
ax = p.ax, size=4, color='k')
p.ax.set_xlabel('');
p.fig.set_size_inches(8,4)
Histogramas¶
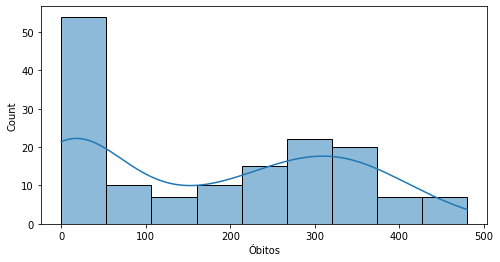
O seaborn constrói histogramas a partir da função histplot, ou da função obsoleta distplot.
Incluímos um estimador de densidade baseado em núcleo Gaussiano (Gaussian kernel) utilizando o argumento kde=True.
fig, ax = plt.subplots(figsize=(8,4))
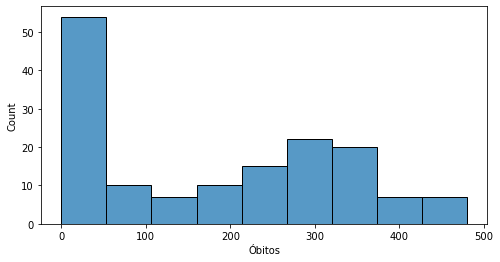
_ = sns.histplot(covid_regioes_diarios_px.query('Região=="Nordeste"')['Óbitos'],kde=True)
Para remover o estimador, selecionamos kde=False.
fig, ax = plt.subplots(figsize=(8,4))
_ = sns.histplot(covid_regioes_diarios_px.query('Região=="Nordeste"')['Óbitos'],kde=False)
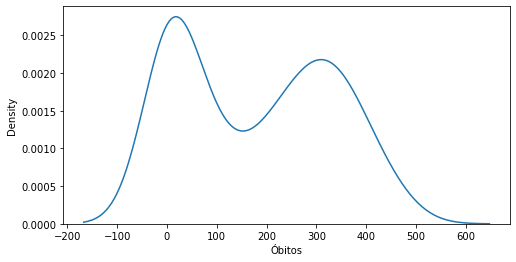
Para plotarmos apenas o estimador de densidade sem o histograma, usamos distplot com a opção hist=False.
fig, ax = plt.subplots(figsize=(8,4))
_ = sns.distplot(covid_regioes_diarios_px.query('Região=="Nordeste"')['Óbitos'],hist=False)
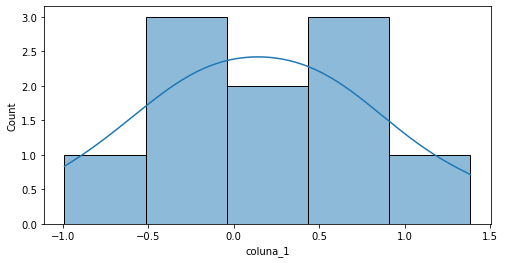
É possível plotar o histograma com o estimador para qualquer série do DataFrame.
fig, ax = plt.subplots(figsize=(8,4))
_ = sns.histplot(df_exemplo['coluna_1'],kde=True)
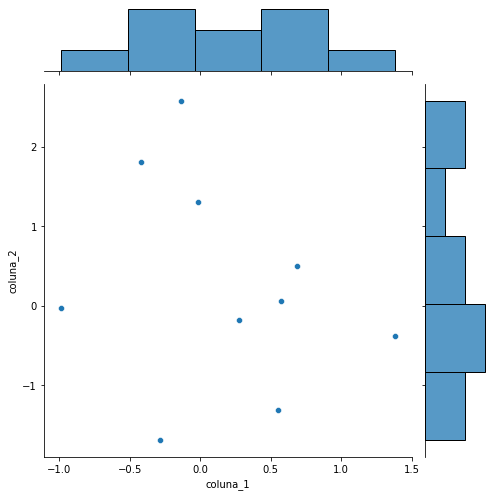
Distribuição conjunta e marginal¶
O histograma permite que verifiquemos a distribuição de uma ou mais variáveis, mas sem levar outras em consideração. Para plotarmos uma distribuição conjunta, bem como a distribuição individual (marginal) de cada variável, podemos utilizar a função jointplot.
_ = sns.jointplot(x = 'coluna_1', y = 'coluna_2', data=df_exemplo, height=7)
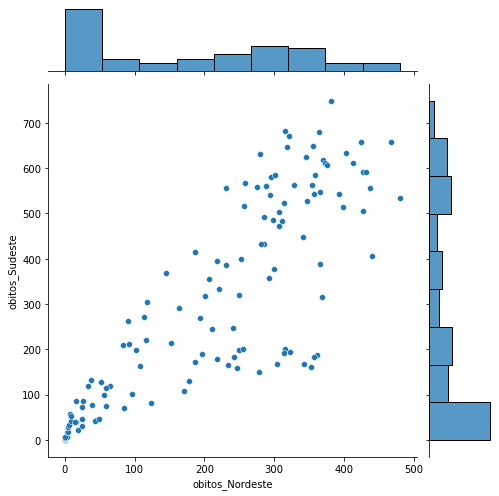
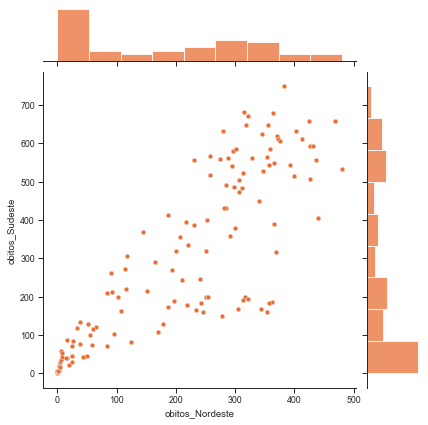
O próximo exemplo usa a função jointplot para plotar distribuições conjuntas relativas ao número de óbitos por Covid-19 por região do Brasil durante uma certa janela temporal.
covid_regioes_diarios = pd.DataFrame()
regioes = covid_BR.query('regiao != "Brasil"')['regiao'].drop_duplicates().array
for regiao in regioes:
temp_series = covid_BR.set_index('data').query('regiao == @regiao')['obitosNovos'].groupby('data').sum()/2
temp_series.name = 'obitos_' + regiao
covid_regioes_diarios = pd.concat([covid_regioes_diarios, temp_series], axis=1)
covid_regioes_diarios.index = pd.to_datetime(covid_regioes_diarios.index)
covid_regioes_diarios.head()
| obitos_Norte | obitos_Nordeste | obitos_Sudeste | obitos_Sul | obitos_Centro-Oeste | |
|---|---|---|---|---|---|
| 2020-02-25 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2020-02-26 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2020-02-27 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2020-02-28 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2020-02-29 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
_ = sns.jointplot(x='obitos_Nordeste', y='obitos_Sudeste',
data = covid_regioes_diarios, height=7)
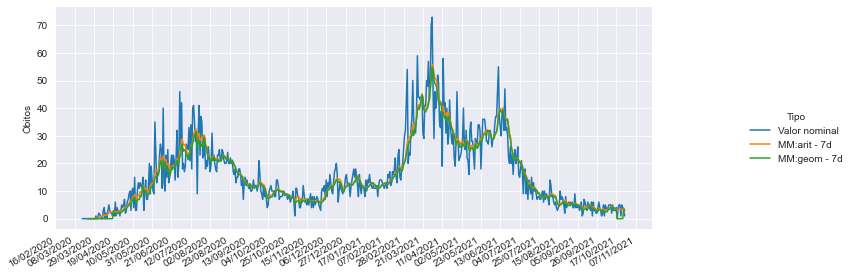
Estilos e cores¶
O seaborn disponibiliza 5 estilos pré-definidos: darkgrid, whitegrid, dark, white e ticks. Vejamos cada um deles.
import matplotlib.dates as mdates
from matplotlib.ticker import FuncFormatter
sns.set_style("darkgrid")
p = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
p.fig.autofmt_xdate()
p.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
p.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
p.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
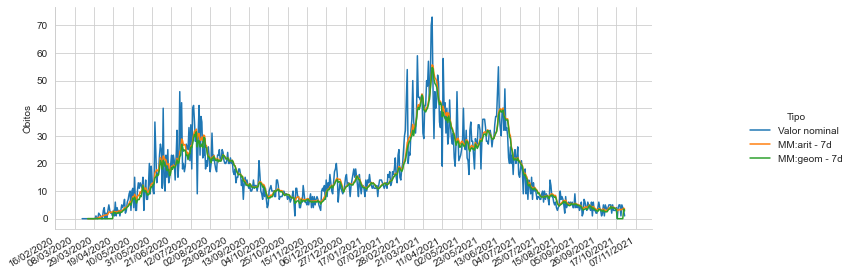
sns.set_style("whitegrid")
p = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
p.fig.autofmt_xdate()
p.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
p.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
p.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
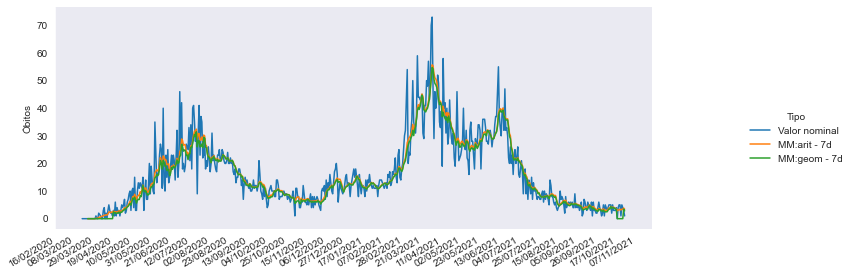
sns.set_style("dark")
p = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
p.fig.autofmt_xdate()
p.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
p.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
p.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
sns.set_style("white")
p = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
p.fig.autofmt_xdate()
p.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
p.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
p.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
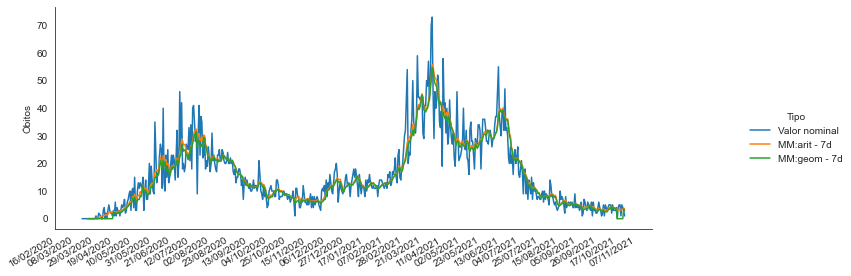
sns.set_style("ticks") # A diferença com o anterior são os "ticks" no eixo x
p = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
p.fig.autofmt_xdate()
p.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
p.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
p.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
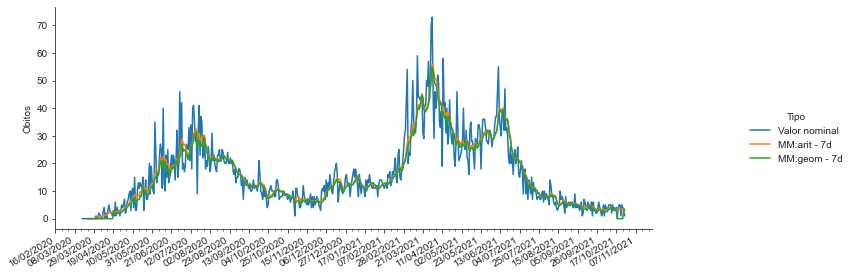
Molduras¶
Utilizamos a função despine para adicionar ou remover molduras em plotagens com o seaborn. Podemos especificar lados para adicionar e remover molduras utilizando os booleanos True e False.
sns.set_style("ticks")
p = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
p.fig.autofmt_xdate()
p.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7))
p.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21))
p.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y'))
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
sns.despine(right=False, top=False)
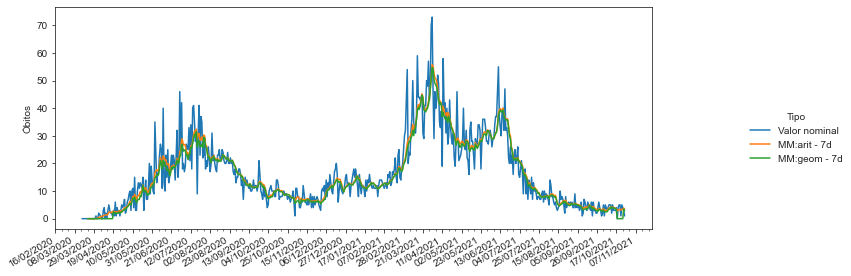
Podemos criar um tipo de “acolchoamento” aumentando a distância entre o gráfico e a moldura utilizando offset. Atribuindo um valor para este argumento, as bordas serão afastadas naturalmente. Um tipo de “corte estético” pode ser adicionado à moldura com trim=False.
sns.set_style("ticks")
p = sns.relplot(x = 'data', y='Óbitos',
hue = 'Tipo',
data=covid_PB_obitos_df.reset_index(),
kind='line')
p.fig.autofmt_xdate()
p.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
p.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
p.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
sns.despine(right=False, top=False, offset=30)
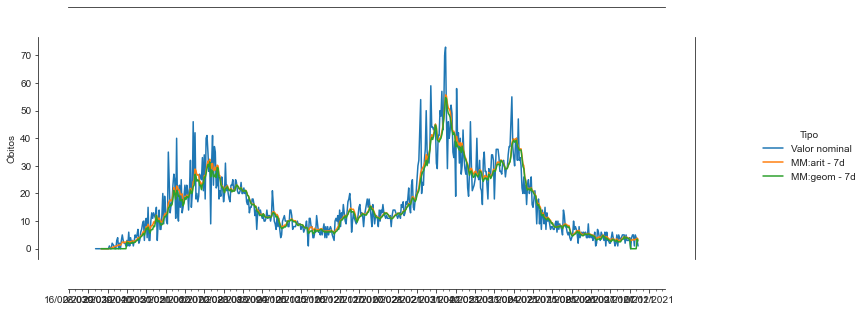
sns.set_style("ticks")
p = sns.relplot(x = 'data', y='Óbitos',
hue = 'Tipo',
data=covid_PB_obitos_df.reset_index(),
kind='line')
p.fig.autofmt_xdate()
p.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
p.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
p.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
sns.despine(offset=30, trim=True)
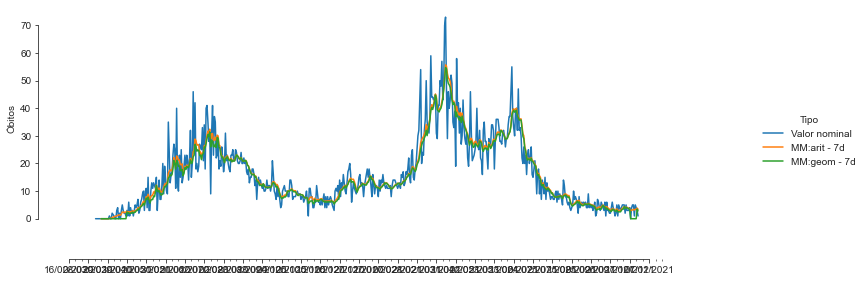
Contextos e escala¶
O seaborn possui contextos pré-definidos que mudam a escala do gráfico para melhor satisfazer a aplicação de interesse. Para definir o contexto, utilizamos a função set_context. Há 4 contextos pré-definidos: paper, notebook, talk e poster.
sns.set_context("poster")
sns.set_style("ticks")
p = sns.relplot(x = 'data', y='Óbitos',
hue = 'Tipo',
data=covid_PB_obitos_df.reset_index(),
kind='line')
p.fig.autofmt_xdate()
p.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
p.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
p.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
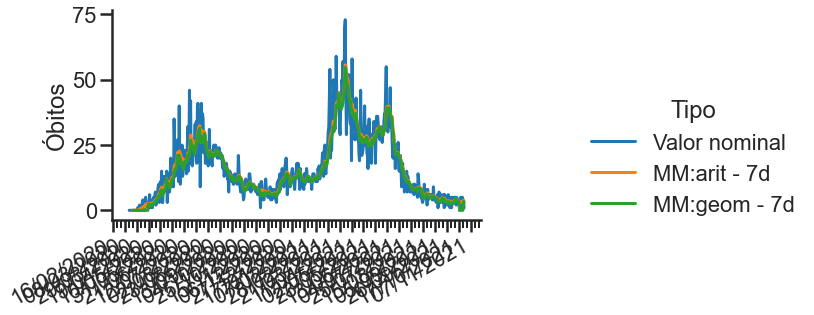
sns.set_context("talk")
sns.set_style("ticks")
p = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
p.fig.autofmt_xdate()
p.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
p.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
p.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
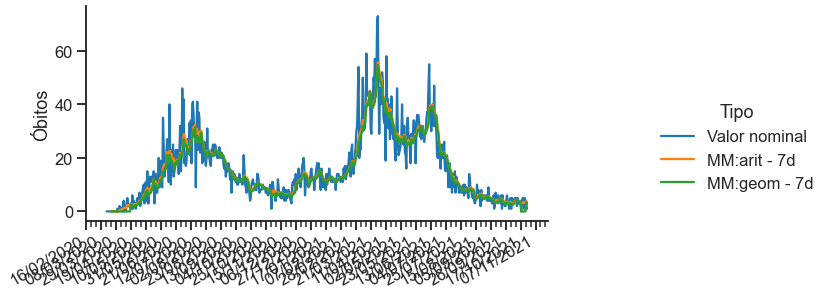
sns.set_context("notebook")
sns.set_style("ticks")
p = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
p.fig.autofmt_xdate()
p.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
p.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
p.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
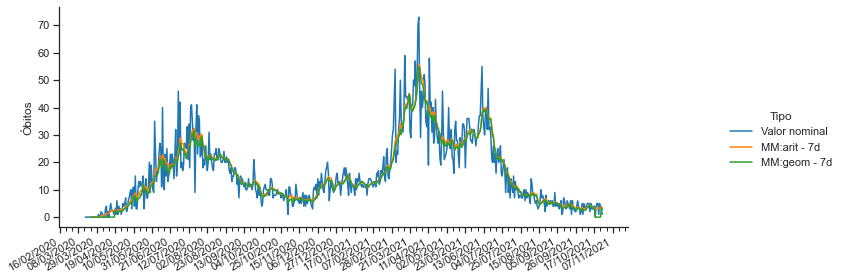
sns.set_context("paper")
sns.set_style("ticks")
p = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
p.fig.autofmt_xdate()
p.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
p.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
p.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
Paletas e mapas de cores¶
É possível personalizar a paleta de cores a ser utilizada ou escolher uma da lista (extremamente extensa) de paletas disponíveis utilizando a função set_palette. Duas formas usuais de selecionar a paleta de cores são:
no escopo de uma instrução
withpor meio da funçãocolor_palette;pelo argumento
palettenas funções de construção gráfica.
A função color_palette() aceita nomes de uma paleta do seaborn (deep, muted, bright, pastel,dark,colorbind), um mapa de cores (colormap) do matplotlib, uma sequência de cores em qualquer formato aceitável pelo matplotlib, entre outras opções.
Note
Para uma lista ampla de paletas, veja a discussão neste post ou a Colormap reference do matplotlib.
Abaixo temos alguns exemplos de paletas nativas do seaborn
sns.color_palette()
sns.color_palette('colorblind')
sns.color_palette('bright')
e de mapas do cores do matplotlib.
sns.color_palette('Greens')
sns.color_palette('turbo')
sns.color_palette('cividis')
Nos exemplos a seguir, plotamos alguns gráficos novamente com paletas diferentes.
# paleta: 'BuPu'
sns.set_context("paper")
sns.set_style("ticks")
p = sns.relplot(x = 'data', y='Óbitos',
hue = 'Tipo',
data=covid_PB_obitos_df.reset_index(), kind='line',
palette = 'BuPu')
p.fig.autofmt_xdate()
p.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
p.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
p.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
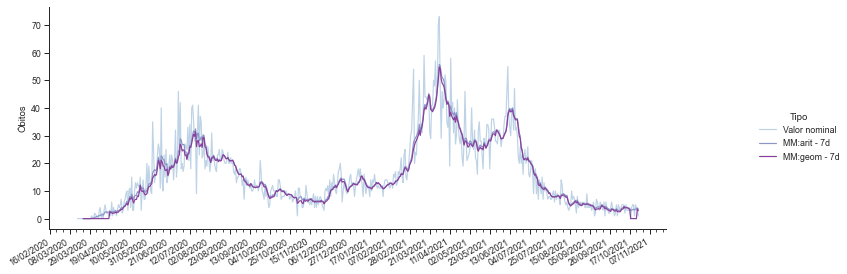
# paleta: 'mako_r'
with sns.color_palette('mako_r'):
p = sns.catplot(x='Região', y='Óbitos',
kind = 'violin',
data=covid_regioes_diarios_px)
p.ax.set_xlabel(''); p.fig.set_size_inches(12,4)
# paleta: icefire_r
sns.set_palette('icefire_r')
_ = sns.jointplot(x='obitos_Nordeste', y='obitos_Sudeste',
data = covid_regioes_diarios,
height=6)
Nota¶
Este capítulo baseia-se nas notas de aula da Profa. Andrea Rocha (CI/UFPB), elaboradas para o mini-curso FMECD.
