Visualização gráfica de dados com seaborn¶
A biblioteca seaborn foi construída em cima do matplotlib e fornece essencialmente duas coisas:
estilo aos gráficos, o que os torna tipicamente muito mais bonitos visualmente;
“agrupa” diversos comandos gráficos do matplotlib dentro de um único comando, o que tipicamente torna simples a realização de gráficos com bastante elementos.
Vamos começar importando as bibliotecas que utilizaremos:
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
Vamos construir e importar os DataFrames que utilizaremos:
serie_Idade = pd.Series({'Ana':20, 'João': 19, 'Maria': 21, 'Pedro': 22, 'Túlio': 20}, name="Idade")
serie_Peso = pd.Series({'Ana':55, 'João': 80, 'Maria': 62, 'Pedro': 67, 'Túlio': 73}, name="Peso")
serie_Altura = pd.Series({'Ana':162, 'João': 178, 'Maria': 162, 'Pedro': 165, 'Túlio': 171}, name="Altura")
dicionario_series_exemplo = {'Idade': serie_Idade, 'Peso': serie_Peso, 'Altura': serie_Altura}
df_dict_series = pd.DataFrame(dicionario_series_exemplo);df_dict_series
| Idade | Peso | Altura | |
|---|---|---|---|
| Ana | 20 | 55 | 162 |
| João | 19 | 80 | 178 |
| Maria | 21 | 62 | 162 |
| Pedro | 22 | 67 | 165 |
| Túlio | 20 | 73 | 171 |
df_exemplo = pd.read_csv('data/exemplo_data.csv', index_col=0)
df_exemplo['coluna_3'] = pd.Series([1,2,3,4,5,6,7,8,np.nan,np.nan],index=df_exemplo.index)
df_exemplo.index = pd.to_datetime(df_exemplo.index)
df_exemplo
| coluna_1 | coluna_2 | coluna_3 | |
|---|---|---|---|
| 2020-01-01 | -0.416092 | 1.810364 | 1.0 |
| 2020-01-02 | -0.137970 | 2.578520 | 2.0 |
| 2020-01-03 | 0.575827 | 0.060866 | 3.0 |
| 2020-01-04 | -0.017367 | 1.299587 | 4.0 |
| 2020-01-05 | 1.384279 | -0.381732 | 5.0 |
| 2020-01-06 | 0.549706 | -1.308789 | 6.0 |
| 2020-01-07 | -0.282296 | -1.688979 | 7.0 |
| 2020-01-08 | -0.989730 | -0.028121 | 8.0 |
| 2020-01-09 | 0.275582 | -0.177659 | NaN |
| 2020-01-10 | 0.685132 | 0.502535 | NaN |
covid_PB = pd.read_csv('https://superset.plataformatarget.com.br/superset/explore_json/?form_data=%7B%22slice_id%22%3A1550%7D&csv=true',
sep=',', index_col=0)
covid_PB.head()
| casosAcumulados | casosNovos | descartados | recuperados | obitosAcumulados | obitosNovos | Letalidade | |
|---|---|---|---|---|---|---|---|
| data | |||||||
| 2021-02-09 | 199706 | 971 | 237369 | 152779 | 4171 | 13 | 0.0209 |
| 2021-02-08 | 198735 | 611 | 237189 | 151879 | 4158 | 12 | 0.0209 |
| 2021-02-07 | 198124 | 664 | 237072 | 151535 | 4146 | 11 | 0.0209 |
| 2021-02-06 | 197460 | 918 | 236774 | 150175 | 4135 | 12 | 0.0209 |
| 2021-02-05 | 196542 | 1060 | 236216 | 150169 | 4123 | 13 | 0.0210 |
covid_BR = pd.read_excel('data/HIST_PAINEL_COVIDBR_25jul2020.xlsx')
covid_BR.head()
| Unnamed: 0 | regiao | estado | municipio | coduf | codmun | codRegiaoSaude | nomeRegiaoSaude | data | semanaEpi | populacaoTCU2019 | casosAcumulado | casosNovos | obitosAcumulado | obitosNovos | Recuperadosnovos | emAcompanhamentoNovos | interior/metropolitana | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-25 | 9 | 210147125 | 0 | 0 | 0 | 0 | NaN | NaN | NaN |
| 1 | 1 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-26 | 9 | 210147125 | 1 | 1 | 0 | 0 | NaN | NaN | NaN |
| 2 | 2 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-27 | 9 | 210147125 | 1 | 0 | 0 | 0 | NaN | NaN | NaN |
| 3 | 3 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-28 | 9 | 210147125 | 1 | 0 | 0 | 0 | NaN | NaN | NaN |
| 4 | 4 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-29 | 9 | 210147125 | 2 | 1 | 0 | 0 | NaN | NaN | NaN |
Gráficos de Linha e de Dispersão¶
Os gráficos de linha e de dispersão podem ser realizados no seaborn com a função relplot.
Para fazer um gráfico de linha escolhemos kind = “line”;
O gráfico de dispersão é o padrão (kind = “scatter”).
Alternativamente podemos utilizar as funções lineplot e scatterplot. Porém, lineplot e scatterplot são apenas eixos e se comportam essencialmente como o matplotlib ao passo que relplot tem diversos elementos da construção de uma figura no matplotlib já pré-configurados (isto é chamado no seaborn de figure-level plot).
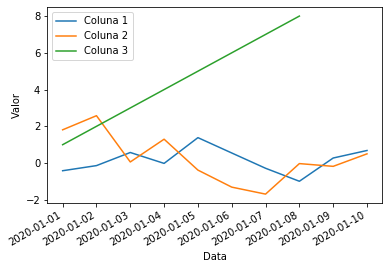
Inicialmente vamos ilustrar com a função lineplot que, como dito, se comporta bastante com as funções do matplotlib.pyplot.
fig, ax = plt.subplots()
ax = sns.lineplot(x="index", y="coluna_1", data=df_exemplo.reset_index(), label = 'Coluna 1')
ax = sns.lineplot(x="index", y="coluna_2", data=df_exemplo.reset_index(), ax = ax, label = 'Coluna 2')
ax = sns.lineplot(x="index", y="coluna_3", data=df_exemplo.reset_index(), ax = ax, label = 'Coluna 3')
ax.set_xlabel('Data')
ax.set_ylabel('Valor')
fig.autofmt_xdate()
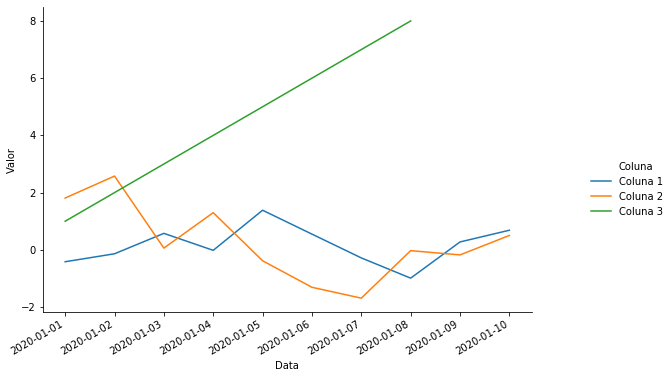
Para utilizar a função relplot precisaremos preparar o banco de dados no mesmo estilo do plotly.express:
df_exemplo_px = pd.DataFrame(df_exemplo['coluna_1']).rename({'coluna_1':'Valor'}, axis=1)
df_exemplo_px['Coluna'] = 'Coluna 1'
df_exemplo_px_temp = pd.DataFrame(df_exemplo['coluna_2']).rename({'coluna_2':'Valor'}, axis=1)
df_exemplo_px_temp['Coluna'] = 'Coluna 2'
df_exemplo_px = pd.concat([df_exemplo_px, df_exemplo_px_temp])
df_exemplo_px_temp = pd.DataFrame(df_exemplo['coluna_3']).rename({'coluna_3':'Valor'}, axis=1)
df_exemplo_px_temp['Coluna'] = 'Coluna 3'
df_exemplo_px = pd.concat([df_exemplo_px, df_exemplo_px_temp])
df_exemplo_px.head()
| Valor | Coluna | |
|---|---|---|
| 2020-01-01 | -0.416092 | Coluna 1 |
| 2020-01-02 | -0.137970 | Coluna 1 |
| 2020-01-03 | 0.575827 | Coluna 1 |
| 2020-01-04 | -0.017367 | Coluna 1 |
| 2020-01-05 | 1.384279 | Coluna 1 |
grafico = sns.relplot(x = 'index', y='Valor', hue = 'Coluna', data=df_exemplo_px.reset_index().dropna(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.set_xlabel('Data')
grafico.fig.set_size_inches(10,5.5)
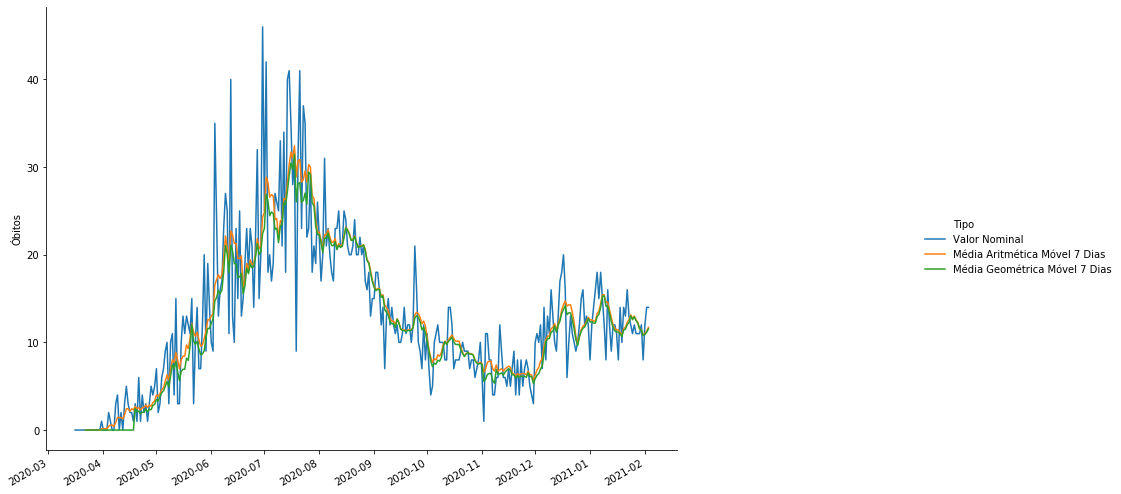
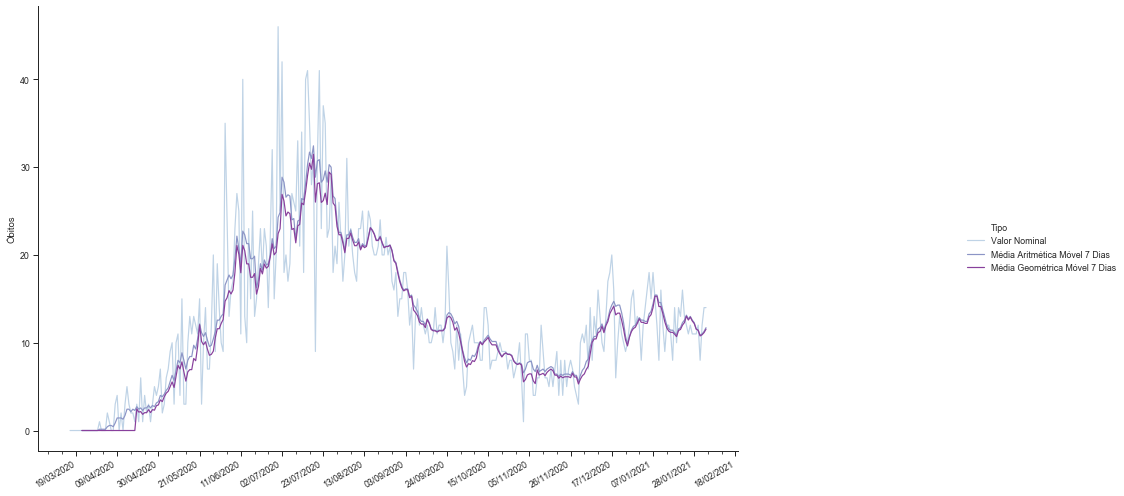
Vamos agora fazer o gráfico de óbitos por COVID-19 na Paraíba juntamente com a média aritmética móvel de 7 dias e com a média geométrica móvel de 7 dias.
A média aritmética tem a desvantagem de linearizar o efeito do crescimento ou decrescimento do número de óbitos, onde sabemos que o efeito é exponencial.
A média geométrica móvel tem a desvantagem de dar zero se o número de óbitos em algum dos dias da janela for zero.
Em geral as duas médias ficam muito próximas.
Utilizaremos o método rolling de uma Serie ou DataFrame do pandas. Este método cria janelas móveis onde podemos aplicar uma função agregadora (tal como média ou média geométrica).
from scipy.stats import gmean # Obtendo a função que calcula média geométrica do scipy
covid_PB_obitos = covid_PB.obitosNovos
covid_PB_obitos = covid_PB_obitos.sort_index()
covid_PB_obitos.name = 'Óbitos'
covid_PB_obitos_df = pd.DataFrame(covid_PB_obitos)
covid_PB_obitos_df['Tipo'] = 'Valor Nominal'
covid_PB_obitos_df_temp = pd.DataFrame(covid_PB_obitos.rolling(7).mean().dropna())
covid_PB_obitos_df_temp['Tipo'] = 'Média Aritmética Móvel 7 Dias'
covid_PB_obitos_df = pd.concat([covid_PB_obitos_df, covid_PB_obitos_df_temp])
covid_PB_obitos_df_temp = pd.DataFrame(covid_PB_obitos.rolling(7).aggregate(gmean).dropna())
covid_PB_obitos_df_temp['Tipo'] = 'Média Geométrica Móvel 7 Dias'
covid_PB_obitos_df = pd.concat([covid_PB_obitos_df, covid_PB_obitos_df_temp])
covid_PB_obitos_df.index = pd.to_datetime(covid_PB_obitos_df.index)
covid_PB_obitos_df.tail()
| Óbitos | Tipo | |
|---|---|---|
| data | ||
| 2021-01-30 | 11.417941 | Média Geométrica Móvel 7 Dias |
| 2021-01-31 | 10.775362 | Média Geométrica Móvel 7 Dias |
| 2021-02-01 | 10.910138 | Média Geométrica Móvel 7 Dias |
| 2021-02-02 | 11.153061 | Média Geométrica Móvel 7 Dias |
| 2021-02-03 | 11.543999 | Média Geométrica Móvel 7 Dias |
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(17,8)
Vamos agora construir um gráfico de dispersão com o conjunto de dados df_exemplo_px:
grafico = sns.relplot(x = 'index', y='Valor', hue = 'Coluna', data=df_exemplo_px.reset_index().dropna())
grafico.fig.autofmt_xdate()
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(10,7)

Infelizmente o seaborn apresenta este problema com datas e a origem do erro não é do seaborn em si e sim como o matplotlib interpreta datas. Mais detalhes em: https://github.com/mwaskom/seaborn/issues/1641
Vamos forçar os limites a ficarem dentro do mínimo (menos um dia) e do máximo (mais um dia):
grafico = sns.relplot(x = 'index', y='Valor', hue = 'Coluna', data=df_exemplo_px.reset_index().dropna())
grafico.ax.set_xlim((df_exemplo_px.reset_index()['index'].min() - pd.DateOffset(days=1)),
(df_exemplo_px.reset_index()['index'].max() + pd.DateOffset(days=1)))
grafico.fig.autofmt_xdate()
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(10,6)
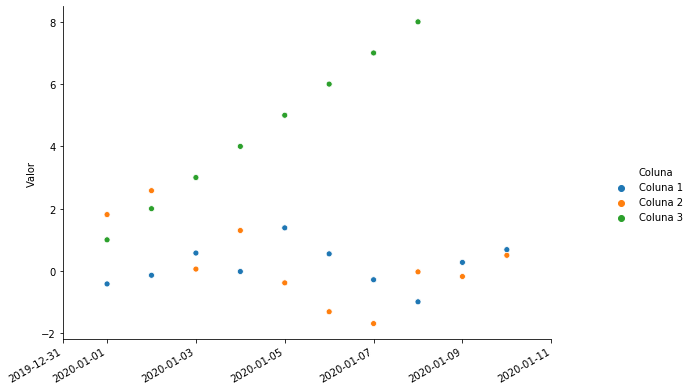
grafico = sns.relplot(x = 'index', y='coluna_1', hue = 'coluna_2', size = 'coluna_3', data=df_exemplo.reset_index().dropna())
grafico.ax.set_xlim((df_exemplo.reset_index()['index'].min() - pd.DateOffset(days=1)),
(df_exemplo.reset_index()['index'].max() + pd.DateOffset(days=1)))
grafico.fig.autofmt_xdate()
grafico.ax.set_xlabel('')
grafico.fig.set_size_inches(10,6)

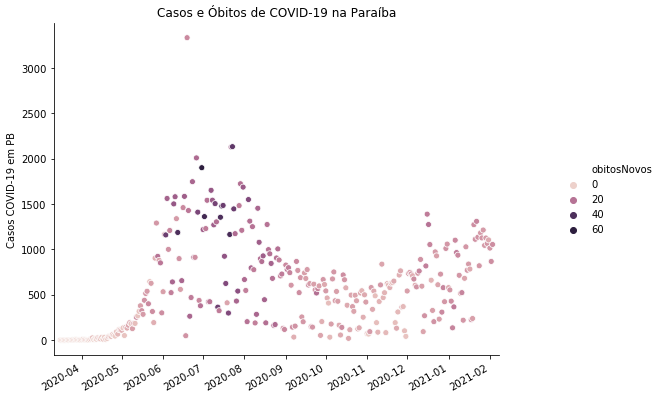
covid_PB_casos_obitos = covid_PB[['obitosNovos', 'casosNovos']].sort_index()
covid_PB_casos_obitos.index = pd.to_datetime(covid_PB_casos_obitos.index)
grafico = sns.relplot(x = 'data', y = 'casosNovos', hue = 'obitosNovos', data=covid_PB_casos_obitos.reset_index())
grafico.ax.set_xlim((covid_PB_casos_obitos.reset_index()['data'].min()-pd.DateOffset(days=5)),
(covid_PB_casos_obitos.reset_index()['data'].max()+ pd.DateOffset(days=5)))
grafico.fig.autofmt_xdate()
grafico.ax.set_xlabel('')
grafico.ax.set_ylabel('Casos COVID-19 em PB')
grafico.ax.set_title('Casos e Óbitos de COVID-19 na Paraíba')
grafico.fig.set_size_inches(10,6)
Gráficos de Dispersão em dados categóricos¶
Neste caso, podemos ter muitos valores repetidos em uma variável, e os gráficos de dispersão podem não ilustrar efetivamente o comportamento dos dados.
Seria interessante gráficos que considerem a repetição de valores dentro de uma mesma categoria.
Alternativas aos Gráficos de Dispersão¶
No caso de termos muitas valores repetidos em uma variável, os gráficos de dispersão deixam de ser eficientes para ilustrar o comportamento dos dados.
Neste sentido, precisamos de gráficos que considerem a repetição de valores.
Obs: Isto acontece quando o eixo horizontal contém variáveis categóricas, e assim, teríamos repetição de valores dentro de uma mesma categoria.
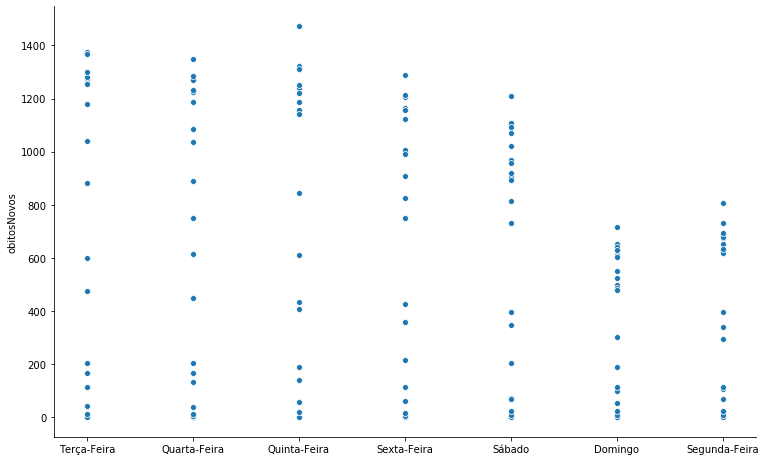
Para estes gráficos vamos utilizar os dados de óbitos por COVID-19 no Brasil. Vamos agrupar o número de óbitos por dia da semana.
covid_BR_obitos = covid_BR.query('regiao == "Brasil"')[['obitosNovos','data']]
covid_BR_obitos.data = pd.to_datetime(covid_BR_obitos.data)
covid_BR_obitos['Dia'] = covid_BR_obitos.data.dt.weekday.map({0:'Segunda-Feira',1:'Terça-Feira',2:'Quarta-Feira',
3:'Quinta-Feira',4:'Sexta-Feira',5:'Sábado', 6:'Domingo'})
covid_BR_obitos = covid_BR_obitos.set_index('data')
covid_BR_obitos
| obitosNovos | Dia | |
|---|---|---|
| data | ||
| 2020-02-25 | 0 | Terça-Feira |
| 2020-02-26 | 0 | Quarta-Feira |
| 2020-02-27 | 0 | Quinta-Feira |
| 2020-02-28 | 0 | Sexta-Feira |
| 2020-02-29 | 0 | Sábado |
| ... | ... | ... |
| 2020-07-21 | 1367 | Terça-Feira |
| 2020-07-22 | 1284 | Quarta-Feira |
| 2020-07-23 | 1311 | Quinta-Feira |
| 2020-07-24 | 1156 | Sexta-Feira |
| 2020-07-25 | 1211 | Sábado |
152 rows × 2 columns
grafico = sns.relplot(x='Dia', y='obitosNovos', data=covid_BR_obitos)
# Se quisermos determinar a ordem do eixo x o relplot não é a função ideal, além disso devido à sobreposição dos dados,
# ela definitivamente não é a ideal para variáveis categóricas.
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(13,7)
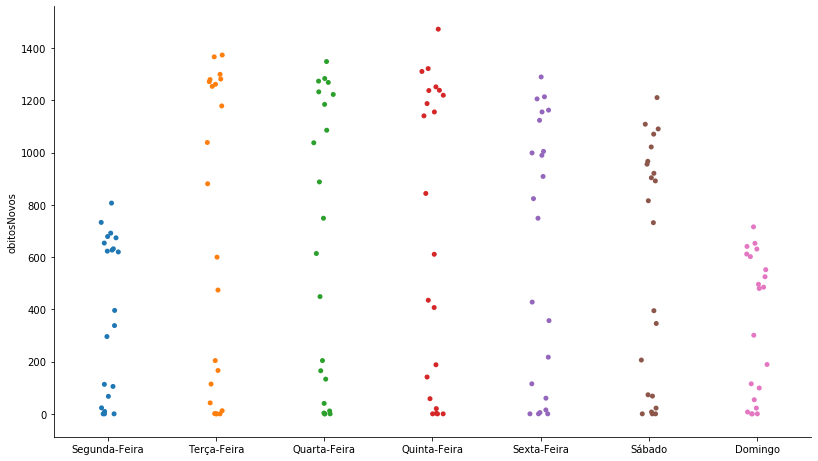
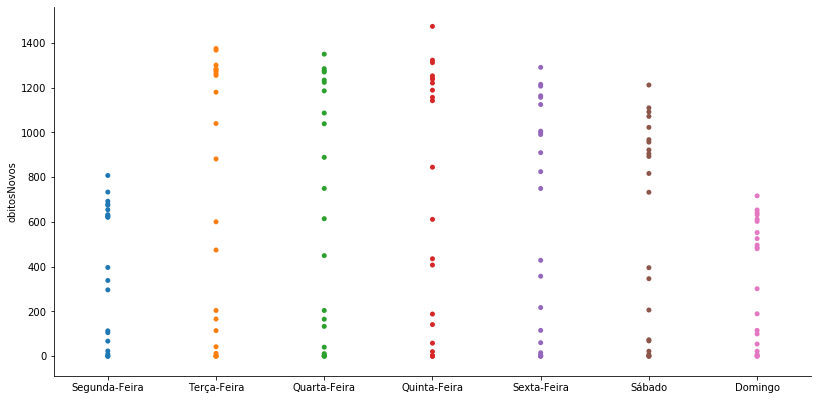
O gráfico stripplot¶
O stripplot é um gráfico de dispersão onde em cada observação é colocado um deslocamento aleatório para evitar a sobreposição e fornecer uma ideia mais precisa da quantidade de dados.
Vamos construir o stripplot através da função catplot. O stripplot é o gráfico padrão do catplot (tem o argumento kind = ‘strip’).
Podemos determinar a ordem das variáveis categóricas com o argumento order.
grafico = sns.catplot(x='Dia', y='obitosNovos', data=covid_BR_obitos, order = ['Segunda-Feira', 'Terça-Feira',
'Quarta-Feira', 'Quinta-Feira', 'Sexta-Feira', 'Sábado', 'Domingo'])
grafico.ax.set_xlabel('');grafico.fig.set_size_inches(13,7)
Se colocarmos jitter=False obtemos o gráfico de dispersão usual (com o detalhe de que podemos definir a ordem dos rótulos do eixo x).
grafico = sns.catplot(x='Dia', y='obitosNovos', jitter = False, data=covid_BR_obitos, order = ['Segunda-Feira', 'Terça-Feira',
'Quarta-Feira', 'Quinta-Feira', 'Sexta-Feira', 'Sábado', 'Domingo'])
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(13,6)
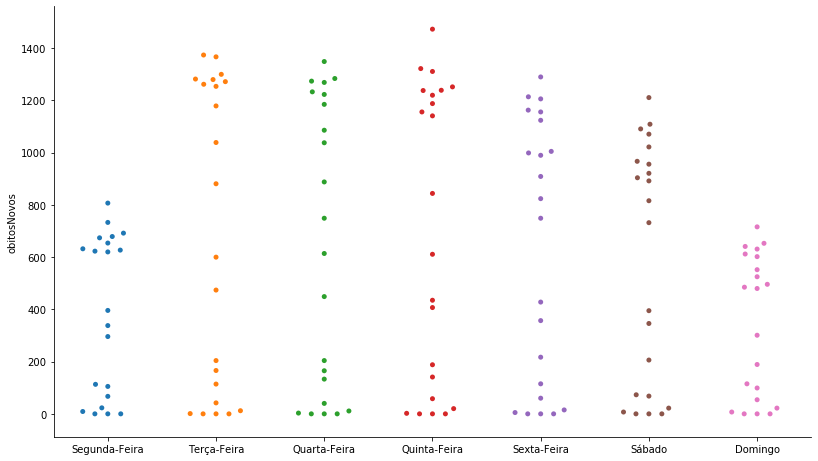
O gráfico swarmplot¶
O swarmplot é um gráfico de dispersão onde, diferentemente do stripplot, nenhum dado pode ficar sobreposto, desta forma também fornece uma ideia mais precisa da quantidade de dados.
Vamos construir o swarmplot através da função catplot com o argumento kind = ‘swarm’.
Como o swarmplot também é um tipo do catplot podemos determinar a ordem das variáveis categóricas com o argumento order.
grafico = sns.catplot(x='Dia', y='obitosNovos', kind = 'swarm', data=covid_BR_obitos, order = ['Segunda-Feira', 'Terça-Feira',
'Quarta-Feira', 'Quinta-Feira', 'Sexta-Feira', 'Sábado', 'Domingo'])
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(13,7)
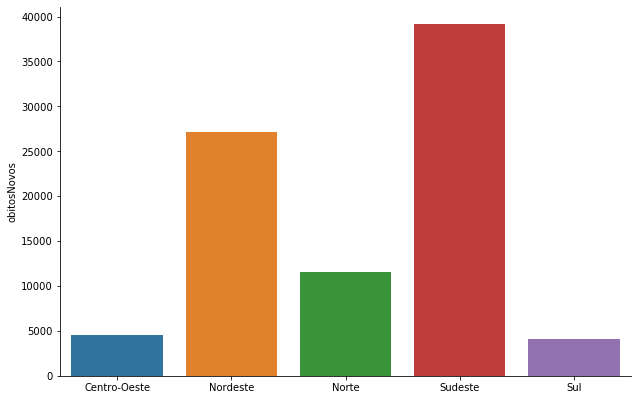
Gráficos de Barras e Colunas¶
Para criar gráficos de barras e colunas com o seaborn utilizaremos a função catplot com o argumento kind=bar.
Se a variável categórica estiver no eixo x o gráfico será de coluna;
Se a variável categórica estiver no eixo y o gráfico será de barra.
covid_Regioes = covid_BR[['regiao','obitosNovos']].groupby('regiao').sum().query('regiao != "Brasil"')/2
grafico = sns.catplot(x='regiao', y='obitosNovos', kind = 'bar',data=covid_Regioes.reset_index())
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(10,6)
covid_Regioes = covid_BR[['regiao','obitosNovos']].groupby('regiao').sum().query('regiao != "Brasil"')/2
grafico = sns.catplot(x='obitosNovos', y='regiao', kind = 'bar',data=covid_Regioes.reset_index())
grafico.ax.set_ylabel('')
grafico.fig.set_size_inches(10,6)
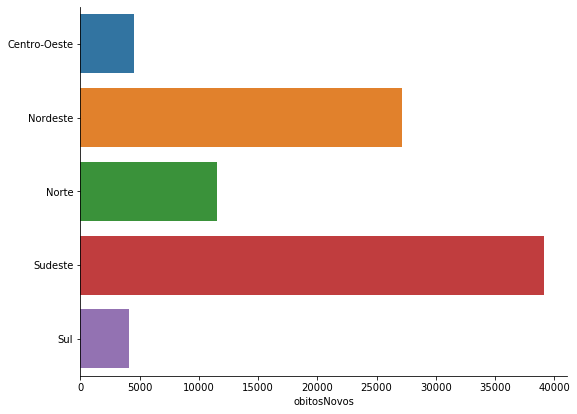
df_dict_series_sns = pd.DataFrame(df_dict_series.Idade).rename({'Idade':'Valor'}, axis=1)
df_dict_series_sns['Dado'] = 'Idade'
df_dict_series_sns_temp = pd.DataFrame(df_dict_series.Altura).rename({'Altura':'Valor'}, axis=1)
df_dict_series_sns_temp['Dado'] = 'Altura'
df_dict_series_sns = pd.concat([df_dict_series_sns, df_dict_series_sns_temp])
df_dict_series_sns_temp = pd.DataFrame(df_dict_series.Peso).rename({'Peso':'Valor'}, axis=1)
df_dict_series_sns_temp['Dado'] = 'Peso'
df_dict_series_sns = pd.concat([df_dict_series_sns, df_dict_series_sns_temp])
df_dict_series_sns
| Valor | Dado | |
|---|---|---|
| Ana | 20 | Idade |
| João | 19 | Idade |
| Maria | 21 | Idade |
| Pedro | 22 | Idade |
| Túlio | 20 | Idade |
| Ana | 162 | Altura |
| João | 178 | Altura |
| Maria | 162 | Altura |
| Pedro | 165 | Altura |
| Túlio | 171 | Altura |
| Ana | 55 | Peso |
| João | 80 | Peso |
| Maria | 62 | Peso |
| Pedro | 67 | Peso |
| Túlio | 73 | Peso |
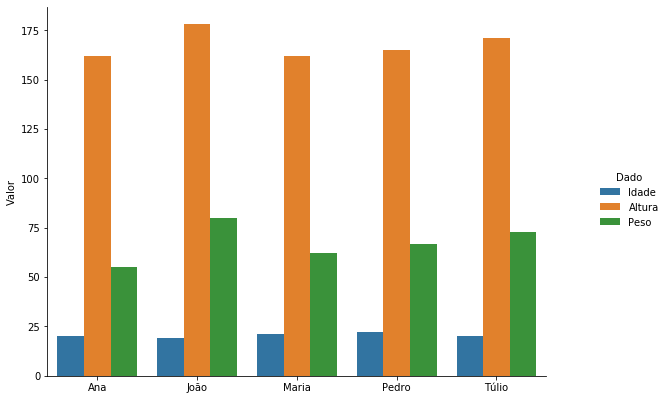
grafico = sns.catplot(x='index', y='Valor', hue='Dado', data = df_dict_series_sns.reset_index(), kind='bar')
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(10,6)
BoxPlot e Alternativas¶
Tanto o BoxPlot quanto as alternativas que apresentaremos aqui (violinplot e boxenplot) fazem parte do catplot.
O boxenplot foi criado por Hadley Wickham (criador do ggplot2 e da maioria dos pacotes do tidyverse do R) e colaboradores e é uma generalização do BoxPlot que apresenta mais quantis. Foi introduzido como letter-value plots: https://vita.had.co.nz/papers/letter-value-plot.html.
O violinplot recebe este nome pois seu gráfico se assemelha a um violino.
Para construir um BoxPlot utiliza-se o argumento kind=‘box’;
Para construir um violinplot utiliza-se o argumento kind=‘violin’;
Para construir um boxenplot (ou letter-value plot) utiliza-se o argumento kind=‘boxen’.
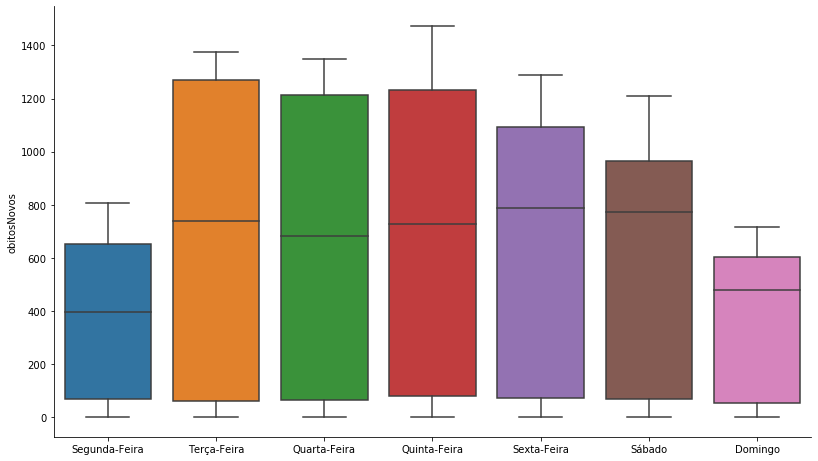
grafico = sns.catplot(x='Dia', y='obitosNovos', kind = 'box', data=covid_BR_obitos, order = ['Segunda-Feira', 'Terça-Feira',
'Quarta-Feira', 'Quinta-Feira', 'Sexta-Feira', 'Sábado', 'Domingo'])
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(13,7)
covid_regioes_diarios_px = covid_BR.set_index(
'data').query('regiao != "Brasil"')[['obitosNovos', 'regiao']].reset_index().rename(
{'obitosNovos':'Óbitos','regiao':'Região','data':'Data'},axis=1)
covid_regioes_diarios_px = covid_regioes_diarios_px.groupby(['Região','Data']).sum()/2
covid_regioes_diarios_px = covid_regioes_diarios_px.reset_index().set_index('Data')
covid_regioes_diarios_px
| Região | Óbitos | |
|---|---|---|
| Data | ||
| 2020-02-25 | Centro-Oeste | 0.0 |
| 2020-02-26 | Centro-Oeste | 0.0 |
| 2020-02-27 | Centro-Oeste | 0.0 |
| 2020-02-28 | Centro-Oeste | 0.0 |
| 2020-02-29 | Centro-Oeste | 0.0 |
| ... | ... | ... |
| 2020-07-21 | Sul | 166.0 |
| 2020-07-22 | Sul | 146.0 |
| 2020-07-23 | Sul | 165.0 |
| 2020-07-24 | Sul | 123.0 |
| 2020-07-25 | Sul | 151.0 |
760 rows × 2 columns
grafico = sns.catplot(x='Região', y='Óbitos', kind = 'box', data=covid_regioes_diarios_px)
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(10,8)
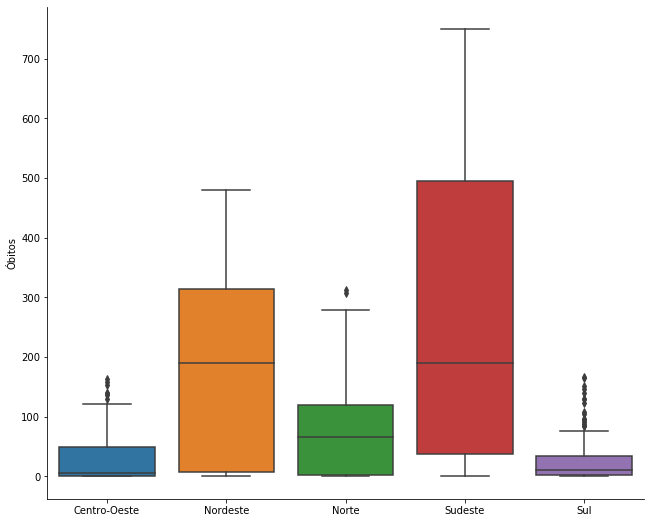
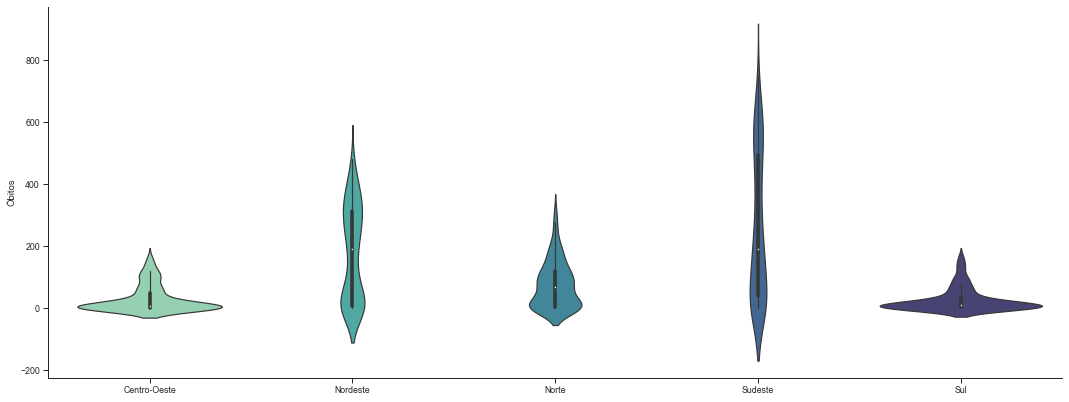
Na presença de muitos outliers, como é o caso do gráfico anterior, é interessante considerar uma alternativa ao BoxPlot.
Vamos ver agora o Boxen Plot (ou letter-value plots). Este plot considera os quantis: …, 0.8%, 1.56%, 3.13%, 6.25%, 12.5%, 25%, 50%, 75%, 87.5%, 93.75%, 96.88%, 98.44%, 99.24%, …
grafico = sns.catplot(x='Região', y='Óbitos', kind = 'boxen', data=covid_regioes_diarios_px)
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(10,8)
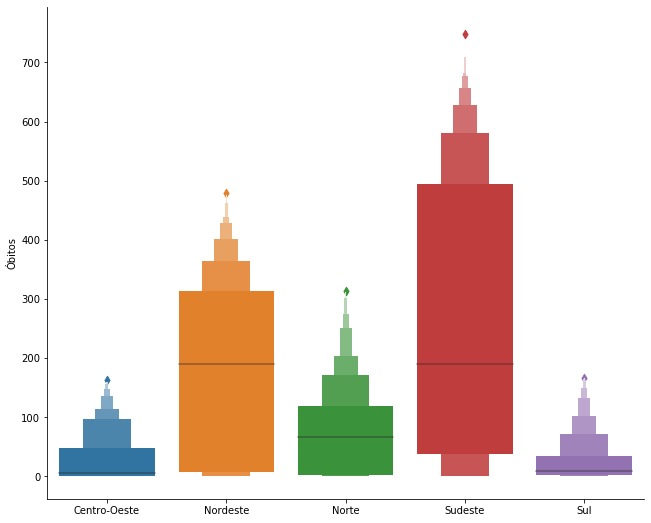
Porém num gráfico sem muitos outliers o Boxen Plot não fica muito diferente do BoxPlot.
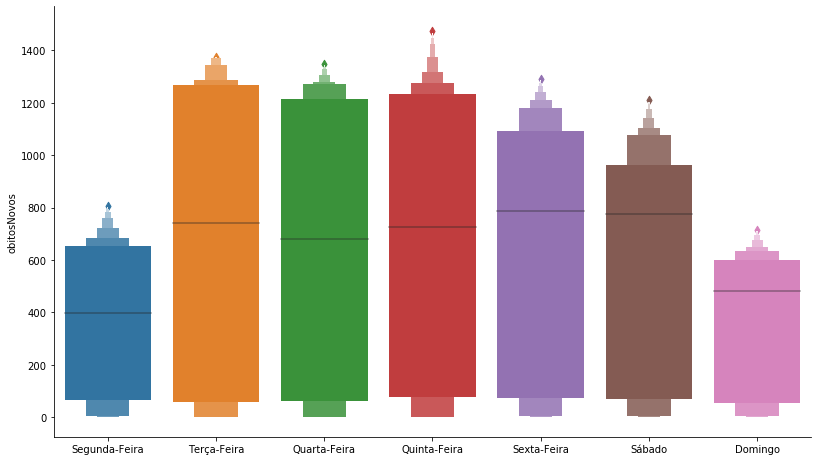
grafico = sns.catplot(x='Dia', y='obitosNovos', kind = 'boxen', data=covid_BR_obitos, order = ['Segunda-Feira', 'Terça-Feira',
'Quarta-Feira', 'Quinta-Feira', 'Sexta-Feira', 'Sábado', 'Domingo'])
grafico.ax.set_xlabel('');grafico.fig.set_size_inches(13,7)
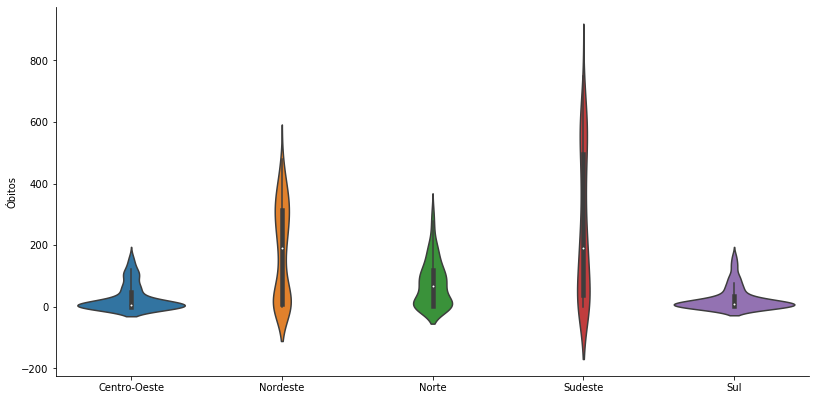
Na presença de muitos outliers também é preferível um violinplot no lugar de um BoxPlot para tornar visível o que está ocorrendo.
grafico = sns.catplot(x='Região', y='Óbitos', kind = 'violin', data=covid_regioes_diarios_px)
grafico.ax.set_xlabel('');grafico.fig.set_size_inches(13,6)
Muitas vezes é interessante sobrepor um violinplot a um swarmplot para evidenciar o comportamento da distribuição dos dados.
grafico = sns.catplot(x='Região', y='Óbitos', kind = 'violin', data=covid_regioes_diarios_px)
sns.swarmplot(x='Região', y='Óbitos', data=covid_regioes_diarios_px, ax = grafico.ax, size=4, color='k') # k = black, b = blue
grafico.ax.set_xlabel('');grafico.fig.set_size_inches(13,6)
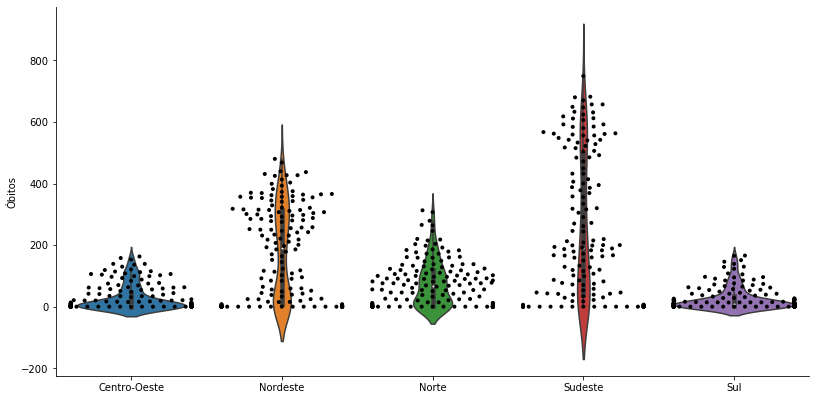
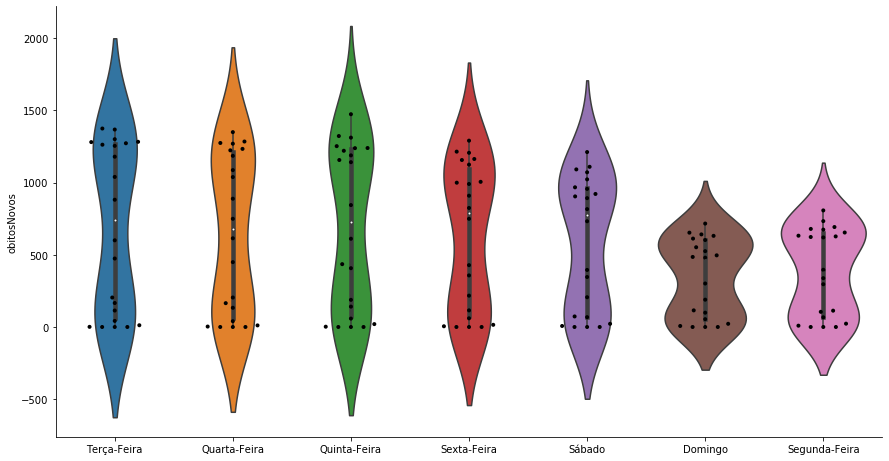
grafico = sns.catplot(x='Dia', y='obitosNovos', kind = 'violin', data=covid_BR_obitos)
sns.swarmplot(x='Dia', y='obitosNovos', data=covid_BR_obitos, ax = grafico.ax, size=4, color='k')
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(15,7)
Histogramas¶
O seaborn constrói histogramas a partir da função distplot e por padrão possui um estimador de densidade baseado no núcleo gaussiano. Podemos retirar o estimador de densidade utilizando o argumento kde=False.
fig, ax = plt.subplots(figsize=(14,10))
_ = sns.distplot(covid_regioes_diarios_px.query('Região=="Nordeste"')['Óbitos'], ax=ax)
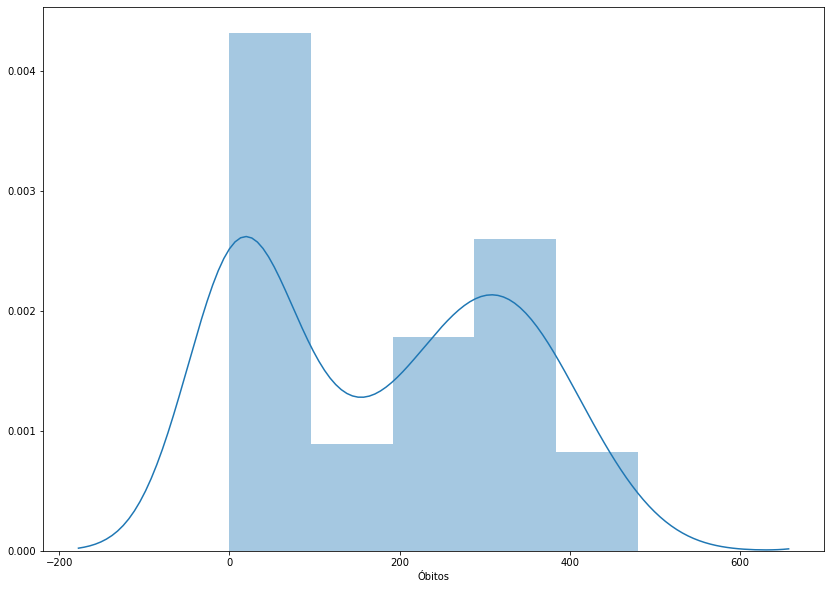
fig, ax = plt.subplots(figsize=(14,10))
_ = sns.distplot(covid_regioes_diarios_px.query('Região=="Nordeste"')['Óbitos'], kde=False, ax=ax)
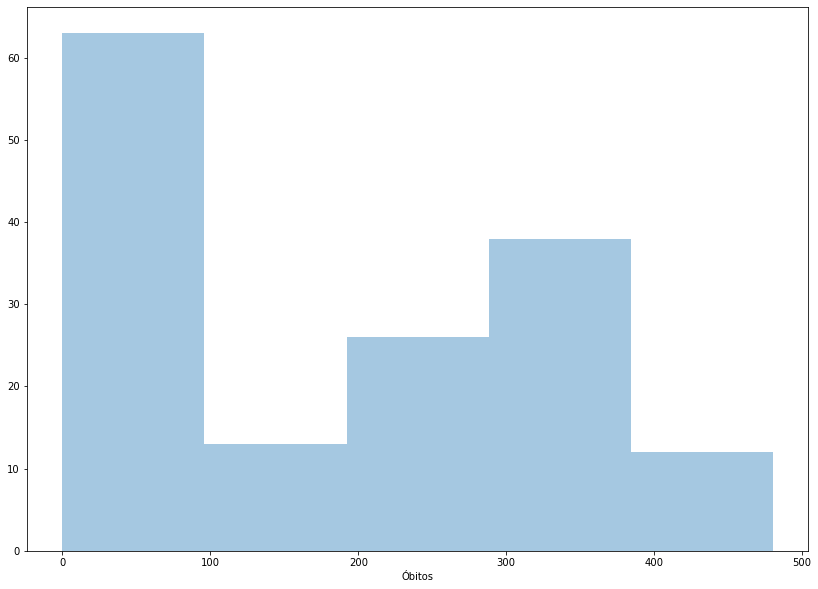
fig, ax = plt.subplots(figsize=(14,10))
_ = sns.distplot(covid_regioes_diarios_px.query('Região=="Nordeste"')['Óbitos'], hist=False)
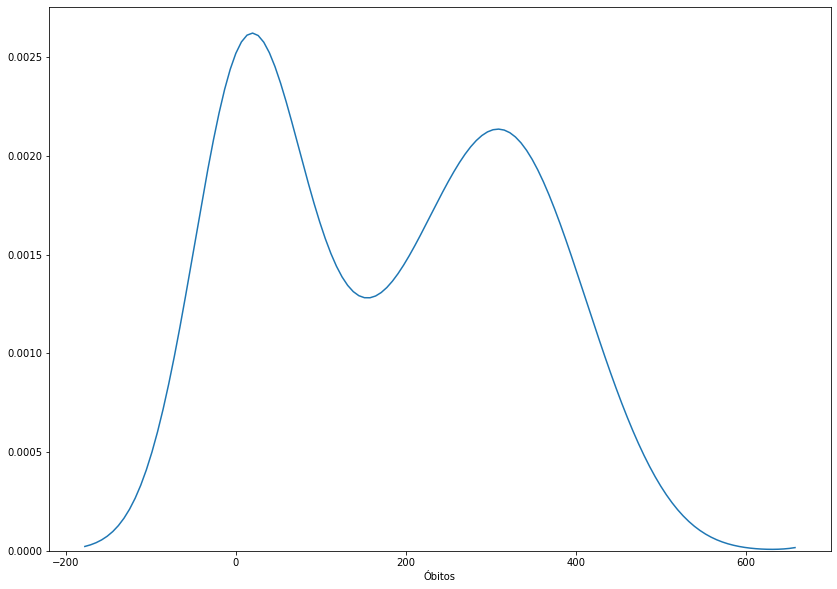
fig, ax = plt.subplots(figsize=(14,10))
_ = sns.distplot(df_exemplo['coluna_1'])
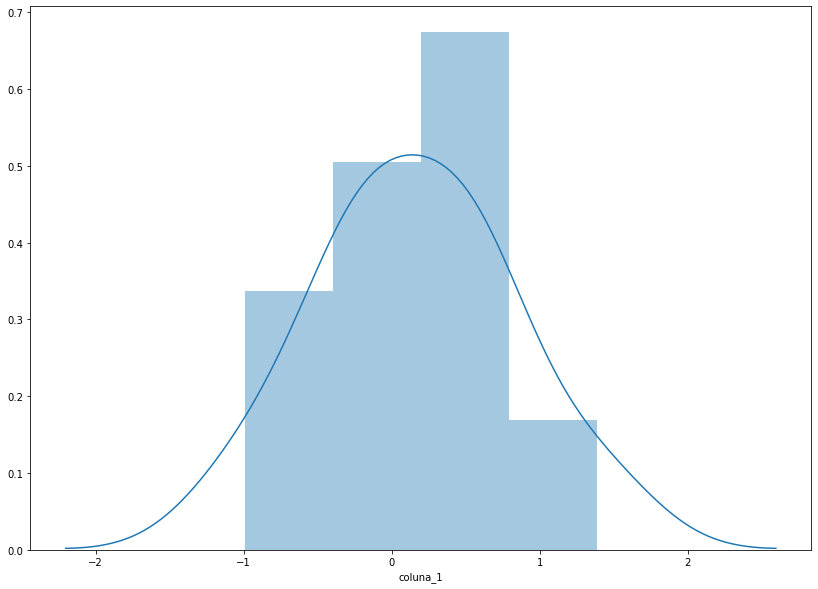
Se quisermos sobrepor dois histogramas, devemos usar a função FacetGrid do seaborn, que permite construir vários gráficos bidimensionais simultaneamente, com a cor sendo dada por uma terceira variável.
Devemos definir qual o DataFrame que iremos trabalhar e qual a variável que queremos que forneça a cor;
Em seguida, devemos aplicar map ao gráfico e definir qual será o tipo de gráfico que queremos, qual variável e quais os parâmetros de interesse.
Para criar gráficos de histogramas com cores, vamos utilizar como base a função criada por lbalazscs no link https://github.com/mwaskom/seaborn/issues/861
Fizemos apenas uma leve modificação para incluir o tamanho e ajeitamos o argumento bins que estava faltando.
def distplot_with_hue(data=None, x=None, hue=None, row=None, col=None, legend=True, size=(10,10), **kwargs):
_, bins = np.histogram(data[x].dropna())
g = sns.FacetGrid(data, hue=hue, row=row, col=col)
g.map(sns.distplot, x, bins=bins, **kwargs)
g.fig.set_size_inches(size)
if legend and (hue is not None) and (hue not in [x, row, col]):
g.add_legend(title=hue)
distplot_with_hue(data=covid_regioes_diarios_px, x='Óbitos', hue='Região', hist=True, size=(18,7))
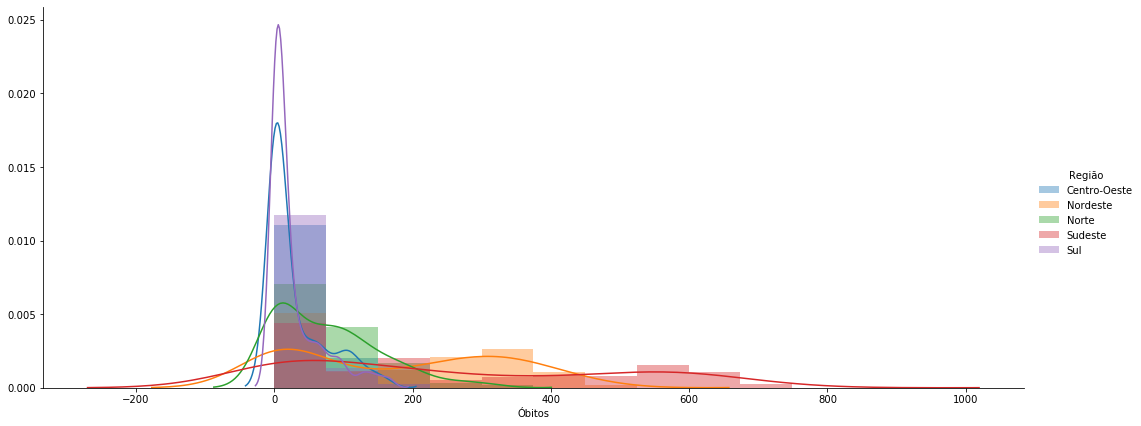
distplot_with_hue(data=covid_regioes_diarios_px, x='Óbitos', hue='Região', hist=True, size=(18,7), kde=False)
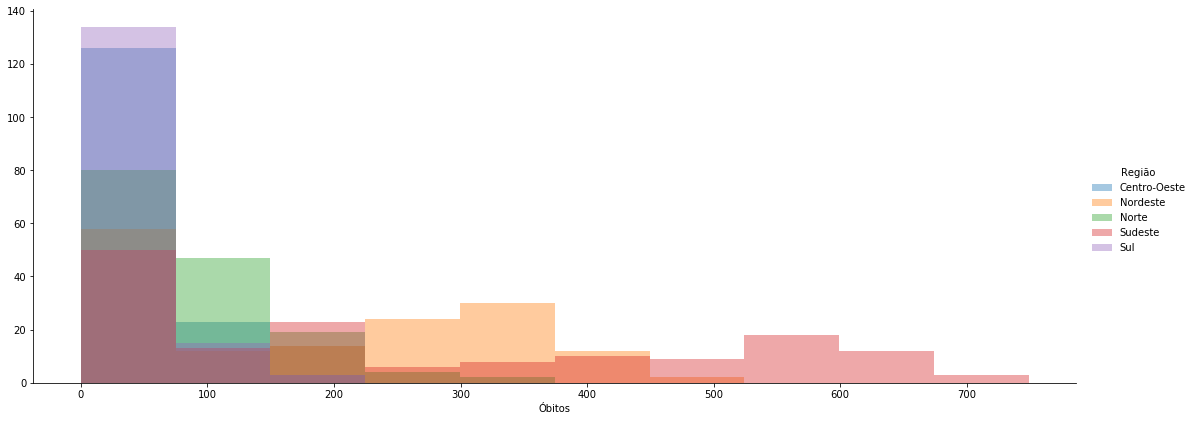
Distribuição Conjunta e Marginais¶
O histograma nos permite verificar a distribuição de uma ou mais variáveis, mas sem levar outras variáveis em conta.
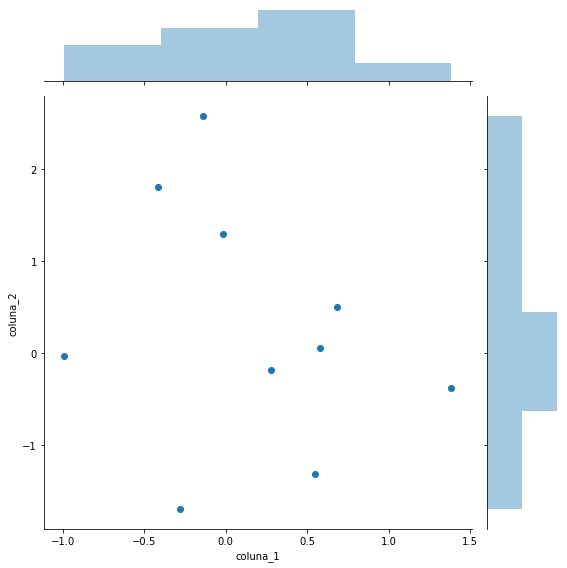
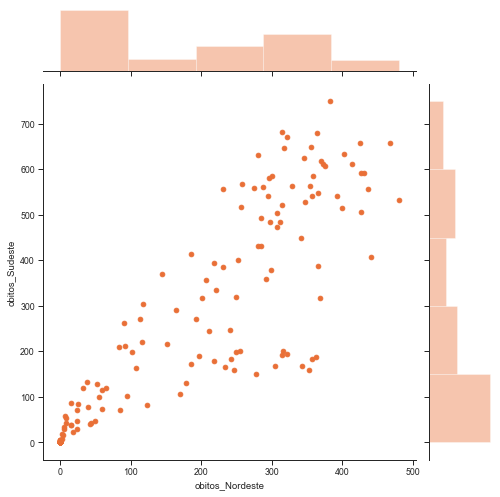
Para verificar como duas variáveis se comportam conjuntamente, isto é, sua distribuição conjunta, assim como suas distribuições individuais (marginais), podemos utilizar a função jointplot do seaborn.
_ = sns.jointplot(x = 'coluna_1', y = 'coluna_2', data=df_exemplo, height=8)
covid_regioes_diarios = pd.DataFrame()
regioes = covid_BR.query('regiao != "Brasil"')['regiao'].drop_duplicates().array
for regiao in regioes:
temp_series = covid_BR.set_index('data').query('regiao == @regiao')['obitosNovos'].groupby('data').sum()/2
temp_series.name = 'obitos_' + regiao
covid_regioes_diarios = pd.concat([covid_regioes_diarios, temp_series], axis=1)
covid_regioes_diarios.index = pd.to_datetime(covid_regioes_diarios.index)
covid_regioes_diarios.head()
| obitos_Norte | obitos_Nordeste | obitos_Sudeste | obitos_Sul | obitos_Centro-Oeste | |
|---|---|---|---|---|---|
| data | |||||
| 2020-02-25 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2020-02-26 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2020-02-27 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2020-02-28 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2020-02-29 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
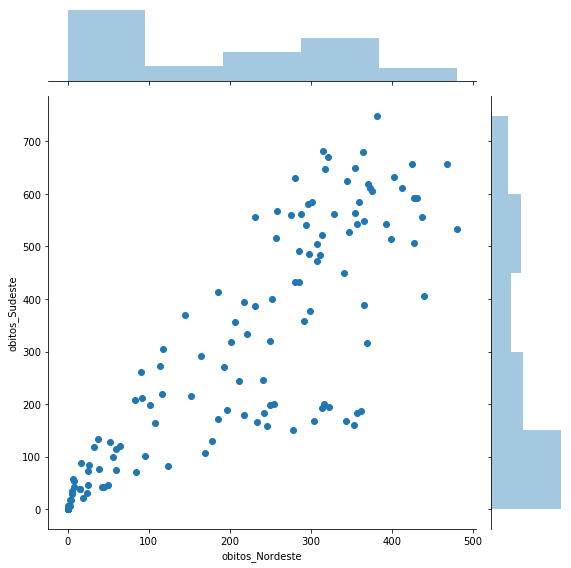
_ = sns.jointplot(x='obitos_Nordeste', y='obitos_Sudeste', data = covid_regioes_diarios, height=8)
Alterando os Estilos e Cores dos Gráficos¶
O seaborn possui 5 estilos pré-definidos: darkgrid, whitegrid, dark, white e ticks.
Vamos ver cada um deles agora.
import matplotlib.dates as mdates
from matplotlib.ticker import FuncFormatter
sns.set_style("whitegrid")
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
grafico.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
grafico.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(20,8)
sns.set_style("darkgrid")
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
grafico.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
grafico.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(20,8)
sns.set_style("dark")
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
grafico.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
grafico.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(20,8)
sns.set_style("white")
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
grafico.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
grafico.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(20,8)
sns.set_style("ticks") # A diferença com o anterior são os "ticks" no eixo x
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
grafico.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
grafico.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(20,8)
Adicionando ou Removendo Molduras¶
Podemos utilizar a função despine para adicionar ou remover molduras num plot do seaborn.
Colocamos qual o lado que queremos adicionar moldura e remover, e colocamos o valor True para remover e False para adicionar.
sns.set_style("ticks")
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
grafico.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
grafico.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(20,8)
sns.despine(right=False, top=False)
Podemos aumentar a distância dos dados com relação à moldura com o argumento offset=dist, onde dist indica a distância que queremos que o gráfico fique da moldura. Ao utilizarmos este argumento as bordas se afastarão naturalmente, devido à distância.
Podemos realizar um “corte” estético na moldura utilizando o argumento trim=True. É uma boa combinação com o offset acima.
sns.set_style("ticks")
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
grafico.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
grafico.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(20,8)
sns.despine(right=False, top=False, offset=30)
sns.set_style("ticks")
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
grafico.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
grafico.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(20,8)
sns.despine(offset=30, trim=True)
Definir o contexto em que o gráfico será utilizado para definir a escala¶
O seaborn possui contextos pré-definidos que mudam a escala do gráfico para melhor satisfazer a aplicação de interesse.
Para definir o contexto, utilizamos a função set_context. A função é bem flexível e para facilitar já vem com 4 contextos pré-definidos: paper, notebook, talk e poster.
sns.set_context("poster")
sns.set_style("ticks")
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
grafico.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
grafico.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(17,8)
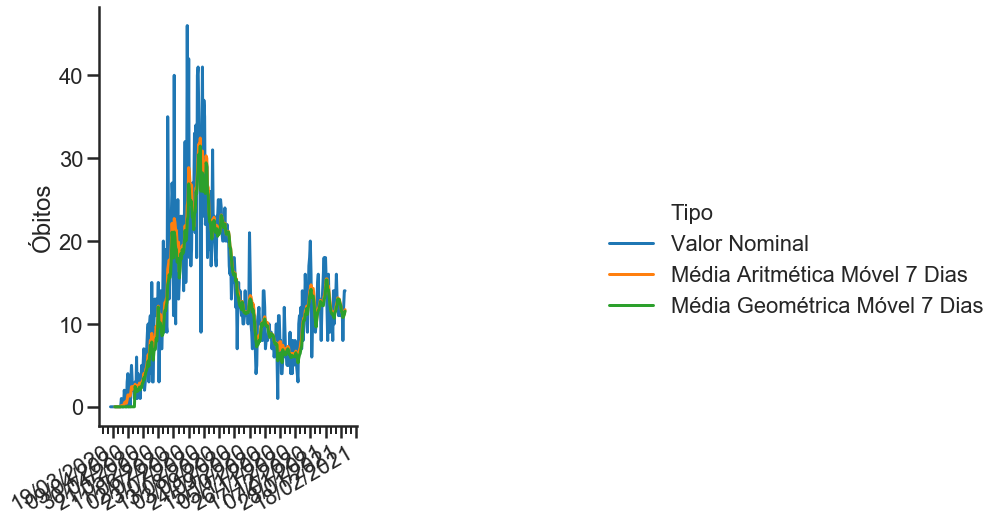
sns.set_context("talk")
sns.set_style("ticks")
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
grafico.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
grafico.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(17,8)
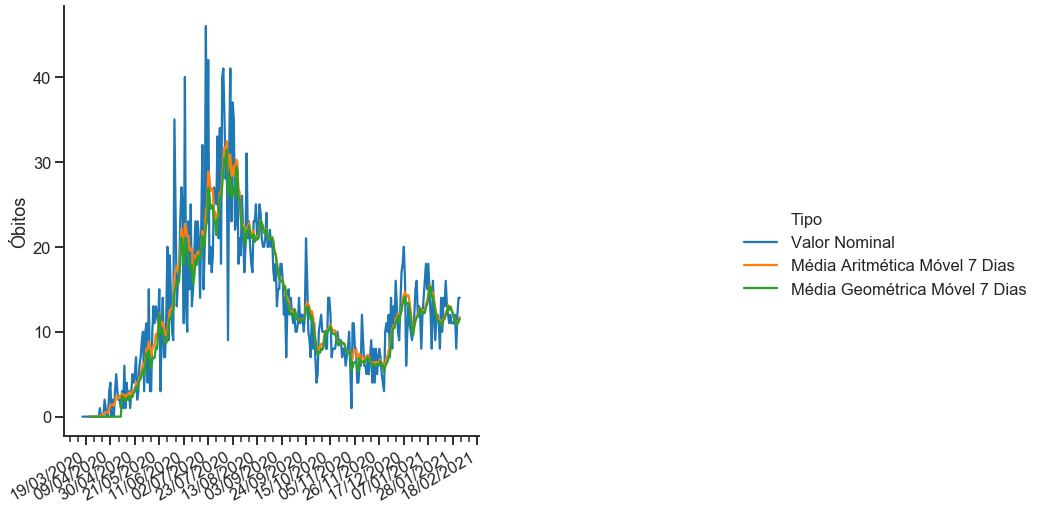
sns.set_context("notebook")
sns.set_style("ticks")
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
grafico.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
grafico.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(17,8)
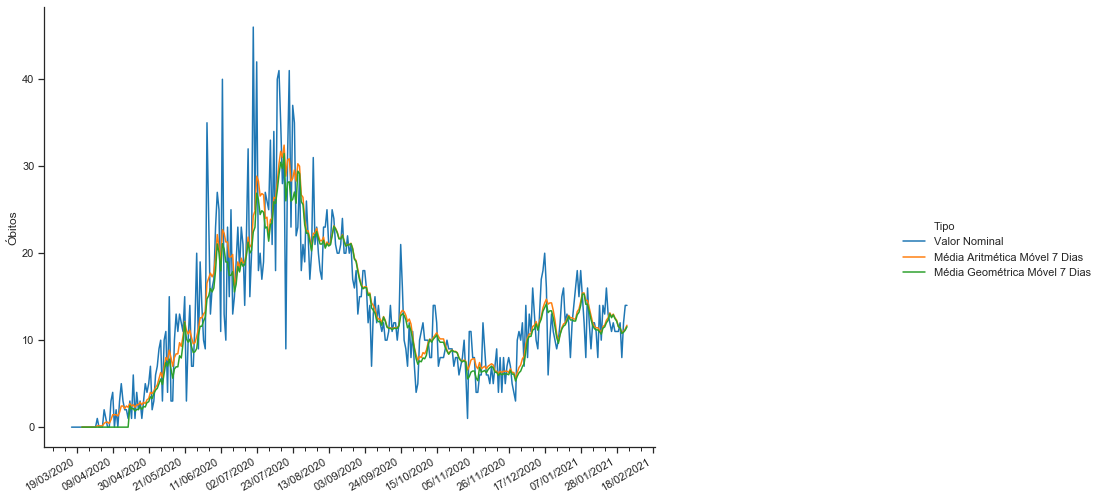
sns.set_context("paper")
sns.set_style("ticks")
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line')
grafico.fig.autofmt_xdate()
grafico.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
grafico.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
grafico.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(17,8)
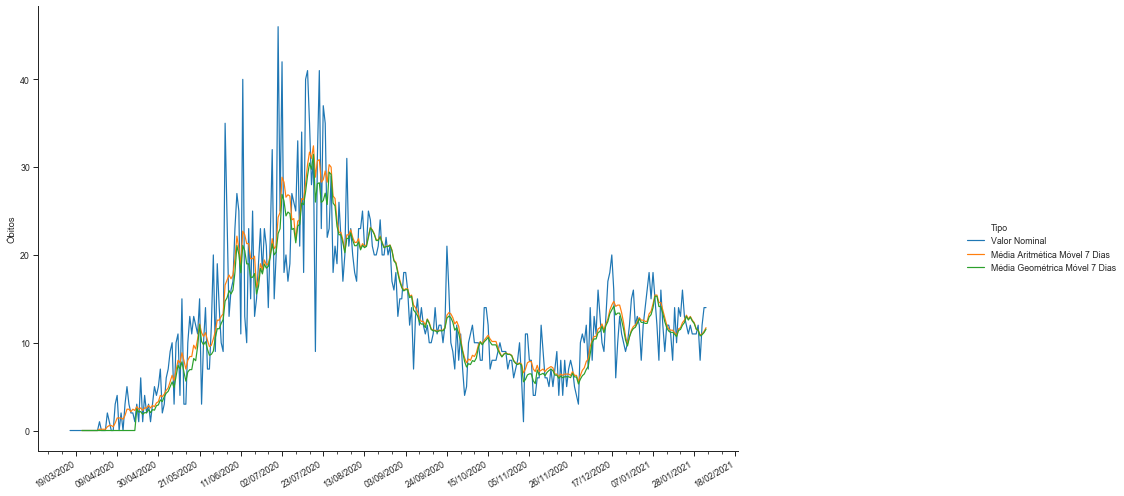
Definindo a paleta de cores a ser utilizada¶
É possível personalizar a paleta a ser utilizada, porém podemos escolher dentre uma lista (extremamente extensa) uma paleta que gostamos e utilizá-la.
Uma lista extensa pode ser encontrada em: https://medium.com/@morganjonesartist/color-guide-to-seaborn-palettes-da849406d44f
Utilizando a função set_palette, é possível definir uma paleta que será utilizada para todos os gráficos.
É possível utilizar uma paleta apenas uma vez, construindo o gráfico dentro de um enunciado with com a função color_palette.
É também possível utilizar uma paleta apenas uma vez utilizando o argumento palette dentro das funções de construções gráficas.
sns.set_context("paper")
sns.set_style("ticks")
grafico = sns.relplot(x = 'data', y='Óbitos', hue = 'Tipo', data=covid_PB_obitos_df.reset_index(), kind='line',
palette = 'BuPu')
grafico.fig.autofmt_xdate()
grafico.ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
grafico.ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
grafico.ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(17,8)
with sns.color_palette('mako_r'):
grafico = sns.catplot(x='Região', y='Óbitos', kind = 'violin', data=covid_regioes_diarios_px)
grafico.ax.set_xlabel(''); grafico.fig.set_size_inches(17,6)
paleta_atual = sns.color_palette() # Obter a paleta de cores sendo utilizada
# Caso haja interesse em guardar este valor, tomar cuidado para não rodar este comando
# enquanto outra paleta esteja sendo utilizada
sns.set_palette('icefire_r')
_ = sns.jointplot(x='obitos_Nordeste', y='obitos_Sudeste', data = covid_regioes_diarios, height=7)
sns.set_palette(paleta_atual) # Voltar para a paleta padrão