Formatação de gráficos com matplotlib¶
Vamos começar refazendo os gráficos que fizemos anteriormente com o método plot dos DataFrames e Series utilizando as funções do matplotlib.pyplot.
O matplotlib transforma os dados em gráficos através de duas componentes: figuras (por exemplo janelas) e eixos (uma região onde os pontos podem ser determinados por meio de coordenadas). Se temos uma figura bidimensional, tipicamente os eixos são x-y, mas podemos ter coordenadas polares também. Se temos uma figura tridimensional, os eixos tipicamente são x-y-z, mas também podemos ter coordenadas esféricas, cilíndricas, etc.
Como as figuras são determinadas pelas posições no plano ou no espaço, utilizamos com mais frequência os eixos de um objeto do matplotlib.
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt # Aqui utilizaremos a biblioteca matplotlib
serie_Idade = pd.Series({'Ana':20, 'João': 19, 'Maria': 21, 'Pedro': 22, 'Túlio': 20}, name="Idade")
serie_Peso = pd.Series({'Ana':55, 'João': 80, 'Maria': 62, 'Pedro': 67, 'Túlio': 73}, name="Peso")
serie_Altura = pd.Series({'Ana':162, 'João': 178, 'Maria': 162, 'Pedro': 165, 'Túlio': 171}, name="Altura")
dicionario_series_exemplo = {'Idade': serie_Idade, 'Peso': serie_Peso, 'Altura': serie_Altura}
df_dict_series = pd.DataFrame(dicionario_series_exemplo);df_dict_series
| Idade | Peso | Altura | |
|---|---|---|---|
| Ana | 20 | 55 | 162 |
| João | 19 | 80 | 178 |
| Maria | 21 | 62 | 162 |
| Pedro | 22 | 67 | 165 |
| Túlio | 20 | 73 | 171 |
df_exemplo = pd.read_csv('06b-exemplo_data.csv', index_col=0)
df_exemplo['coluna_3'] = pd.Series([1,2,3,4,5,6,7,8,np.nan,np.nan],index=df_exemplo.index)
df_exemplo
| coluna_1 | coluna_2 | coluna_3 | |
|---|---|---|---|
| 2020-01-01 | -0.416092 | 1.810364 | 1.0 |
| 2020-01-02 | -0.137970 | 2.578520 | 2.0 |
| 2020-01-03 | 0.575827 | 0.060866 | 3.0 |
| 2020-01-04 | -0.017367 | 1.299587 | 4.0 |
| 2020-01-05 | 1.384279 | -0.381732 | 5.0 |
| 2020-01-06 | 0.549706 | -1.308789 | 6.0 |
| 2020-01-07 | -0.282296 | -1.688979 | 7.0 |
| 2020-01-08 | -0.989730 | -0.028121 | 8.0 |
| 2020-01-09 | 0.275582 | -0.177659 | NaN |
| 2020-01-10 | 0.685132 | 0.502535 | NaN |
covid_PB = pd.read_csv('https://superset.plataformatarget.com.br/superset/explore_json/?form_data=%7B%22slice_id%22%3A1550%7D&csv=true',
sep=',', index_col=0)
covid_PB.head()
| casosAcumulados | casosNovos | descartados | recuperados | obitosAcumulados | obitosNovos | Letalidade | |
|---|---|---|---|---|---|---|---|
| data | |||||||
| 2021-02-03 | 194519 | 1054 | 234902 | 149248 | 4096 | 14 | 0.0211 |
| 2021-02-02 | 193465 | 867 | 234366 | 149242 | 4082 | 14 | 0.0211 |
| 2021-02-01 | 192598 | 1014 | 234215 | 149235 | 4068 | 12 | 0.0211 |
| 2021-01-31 | 191584 | 1104 | 234198 | 148856 | 4056 | 8 | 0.0212 |
| 2021-01-30 | 190480 | 1065 | 233698 | 148722 | 4048 | 12 | 0.0213 |
covid_BR = pd.read_excel("06b-HIST_PAINEL_COVIDBR_18jul2020.xlsx")
covid_BR.head()
| Unnamed: 0 | regiao | estado | municipio | coduf | codmun | codRegiaoSaude | nomeRegiaoSaude | data | semanaEpi | populacaoTCU2019 | casosAcumulado | casosNovos | obitosAcumulado | obitosNovos | Recuperadosnovos | emAcompanhamentoNovos | interior/metropolitana | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-25 | 9 | 210147125 | 0 | 0 | 0 | 0 | NaN | NaN | NaN |
| 1 | 1 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-26 | 9 | 210147125 | 1 | 1 | 0 | 0 | NaN | NaN | NaN |
| 2 | 2 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-27 | 9 | 210147125 | 1 | 0 | 0 | 0 | NaN | NaN | NaN |
| 3 | 3 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-28 | 9 | 210147125 | 1 | 0 | 0 | 0 | NaN | NaN | NaN |
| 4 | 4 | Brasil | NaN | NaN | 76 | NaN | NaN | NaN | 2020-02-29 | 9 | 210147125 | 2 | 1 | 0 | 0 | NaN | NaN | NaN |
Gráfico de Linhas¶
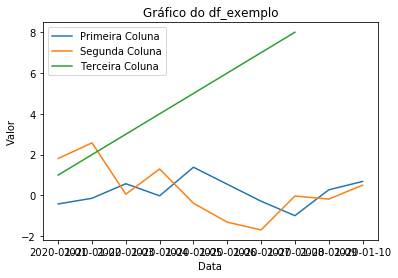
fig, ax = plt.subplots() # Este comando cria uma figura com um eixo
ax.plot(df_exemplo.index, df_exemplo['coluna_1'], label = 'Primeira Coluna') # Inserimos a linha relativa à coluna 1
ax.plot(df_exemplo.index, df_exemplo['coluna_2'], label = 'Segunda Coluna') # Inserimos a linha relativa à coluna 2
ax.plot(df_exemplo.index, df_exemplo['coluna_3'], label = 'Terceira Coluna') # Inserimos a linha relativa à coluna 3
ax.set_xlabel('Data') # Rótulo do eixo x
ax.set_ylabel('Valor') # Rótulo do eixo y
ax.set_title("Gráfico do df_exemplo")
ax.legend()
<matplotlib.legend.Legend at 0x7fa2736a9090>
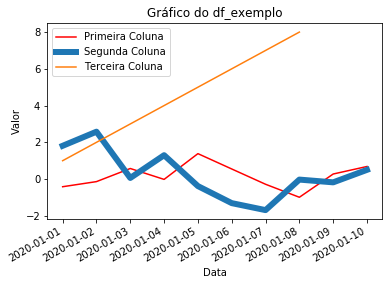
fig, ax = plt.subplots() # Este comando cria uma figura com um eixo
ax.plot(df_exemplo.index, df_exemplo['coluna_1'], label = 'Primeira Coluna',
color = 'red') # Inserimos a linha relativa à coluna 1, definimos a cor vermelha
ax.plot(df_exemplo.index, df_exemplo['coluna_2'],
label = 'Segunda Coluna', linewidth=6.0) # Inserimos a linha relativa à coluna 2 e aumentamos a grossura da linha
ax.plot(df_exemplo.index, df_exemplo['coluna_3'], label = 'Terceira Coluna') # Inserimos a linha relativa à coluna 3
ax.set_xlabel('Data') # Rótulo do eixo x
ax.set_ylabel('Valor') # Rótulo do eixo y
ax.set_title("Gráfico do df_exemplo")
ax.legend()
fig.autofmt_xdate()
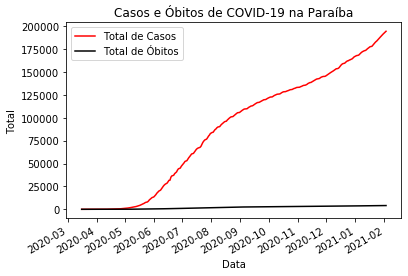
covid_PB.index = pd.to_datetime(covid_PB.index)
covid_PB_casos_obitos = covid_PB[['casosAcumulados', 'obitosAcumulados']].sort_index()
fig, ax = plt.subplots()
ax.plot(covid_PB_casos_obitos.index, covid_PB_casos_obitos['casosAcumulados'], label = 'Total de Casos',
color = 'red')
ax.plot(covid_PB_casos_obitos.index, covid_PB_casos_obitos['obitosAcumulados'],
label = 'Total de Óbitos', color = 'black')
ax.set_xlabel('Data') # Rótulo do eixo x
ax.set_ylabel('Total') # Rótulo do eixo y
ax.set_title("Casos e Óbitos de COVID-19 na Paraíba")
ax.legend()
fig.autofmt_xdate()
Podemos alterar a apresentação das datas utilizando o subpacote dates do matplotlib.
import matplotlib.dates as mdates
fig, ax = plt.subplots()
ax.plot(covid_PB_casos_obitos.index, covid_PB_casos_obitos['casosAcumulados'], label = 'Total de Casos', color = 'red')
ax.plot(covid_PB_casos_obitos.index, covid_PB_casos_obitos['obitosAcumulados'], label = 'Total de Óbitos', color = 'black')
ax.set_xlabel('Data') # Rótulo do eixo x
ax.set_ylabel('Total') # Rótulo do eixo y
ax.set_title("Casos e Óbitos de COVID-19 na Paraíba")
ax.legend()
ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
fig.autofmt_xdate()
Vamos agora alterar o formato dos números do eixo y. Para tanto iremos definir uma função para realizar a formatação e utilizaremos a função FuncFormatter do subpacote matplotlib.ticker.
from matplotlib.ticker import FuncFormatter
def inserir_mil(x, pos):
return '%1i mil' % (x*1e-3) if x != 0 else 0
fig, ax = plt.subplots()
ax.plot(covid_PB_casos_obitos.index, covid_PB_casos_obitos['casosAcumulados'], label = 'Total de Casos', color = 'red')
ax.plot(covid_PB_casos_obitos.index, covid_PB_casos_obitos['obitosAcumulados'], label = 'Total de Óbitos', color = 'black')
ax.set_xlabel('Data') # Rótulo do eixo x
ax.set_ylabel('Total') # Rótulo do eixo y
ax.set_title("Casos e Óbitos de COVID-19 na Paraíba")
ax.legend()
ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
fig.autofmt_xdate()
ax.yaxis.set_major_formatter(FuncFormatter(inserir_mil))
covid_regioes = pd.DataFrame()
regioes = covid_BR.query('regiao != "Brasil"')['regiao'].drop_duplicates().array
for regiao in regioes:
temp_series = covid_BR.set_index('data').query('regiao == @regiao')['obitosAcumulado'].groupby('data').sum()/2
#Obs.: Utilizamos @ na frente do nome da variável para utilizar o valor da variável no query.
#Obs.: Dividimos por 2, pois os óbitos estão sendo contados duas vezes,
#uma para quando codmun == nan e outra quando não é nulo
temp_series.name = 'obitos_' + regiao
covid_regioes = pd.concat([covid_regioes, temp_series], axis=1)
covid_regioes.index = pd.to_datetime(covid_regioes.index)
covid_regioes
| obitos_Norte | obitos_Nordeste | obitos_Sudeste | obitos_Sul | obitos_Centro-Oeste | |
|---|---|---|---|---|---|
| data | |||||
| 2020-02-25 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2020-02-26 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2020-02-27 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2020-02-28 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 2020-02-29 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| ... | ... | ... | ... | ... | ... |
| 2020-07-14 | 10628.0 | 23925.0 | 33718.0 | 2740.0 | 3122.0 |
| 2020-07-15 | 10693.0 | 24272.0 | 34246.0 | 2870.0 | 3285.0 |
| 2020-07-16 | 10790.0 | 24645.0 | 34857.0 | 2975.0 | 3421.0 |
| 2020-07-17 | 10911.0 | 24902.0 | 35374.0 | 3104.0 | 3560.0 |
| 2020-07-18 | 10972.0 | 25194.0 | 35732.0 | 3199.0 | 3675.0 |
145 rows × 5 columns
fig, ax = plt.subplots()
ax.plot(covid_regioes.index, covid_regioes['obitos_Norte'], label = 'Norte')
ax.plot(covid_regioes.index, covid_regioes['obitos_Nordeste'], label = 'Nordeste')
ax.plot(covid_regioes.index, covid_regioes['obitos_Sudeste'], label = 'Sudeste')
ax.plot(covid_regioes.index, covid_regioes['obitos_Sul'], label = 'Sul')
ax.plot(covid_regioes.index, covid_regioes['obitos_Centro-Oeste'], label = 'Centro-Oeste')
ax.set_xlabel('Data') # Rótulo do eixo x
ax.set_ylabel('Total de Óbitos') # Rótulo do eixo y
ax.set_title("Óbitos de COVID-19 nas regiões do Brasil")
ax.legend()
ax.xaxis.set_minor_locator(mdates.DayLocator(interval=7)) #Intervalo entre os tracinhos
ax.xaxis.set_major_locator(mdates.DayLocator(interval=21)) #Intervalo entre as datas
ax.xaxis.set_major_formatter(mdates.DateFormatter('%d/%m/%Y')) #Formato da data
fig.autofmt_xdate()
ax.yaxis.set_major_formatter(FuncFormatter(inserir_mil))
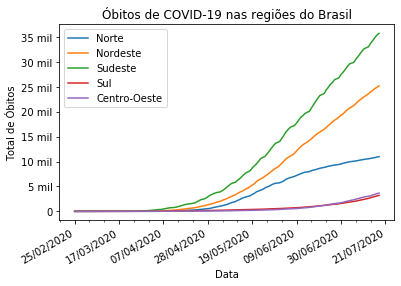
Gráfico de Colunas e de Linhas¶
covid_Regioes = covid_BR[['regiao','obitosNovos']].groupby('regiao').sum().query('regiao != "Brasil"')/2
fig, ax = plt.subplots()
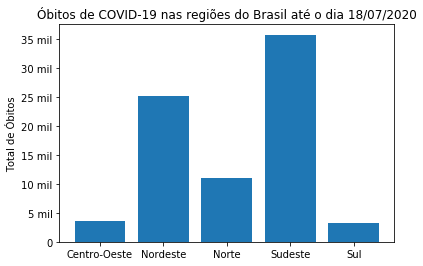
ax.bar(covid_Regioes.index, covid_Regioes['obitosNovos'])
ax.yaxis.set_major_formatter(FuncFormatter(inserir_mil))
ax.set_ylabel('Total de Óbitos') # Rótulo do eixo y
ax.set_title("Óbitos de COVID-19 nas regiões do Brasil até o dia 18/07/2020")
Text(0.5, 1.0, 'Óbitos de COVID-19 nas regiões do Brasil até o dia 18/07/2020')
Podemos inserir o total de cada região em cima do retângulo correspondente. Para tanto, utilizaremos a seguinte função disponível na página do matplotlib:
def autolabel(rects):
"""Attach a text label above each bar in *rects*, displaying its height."""
for rect in rects:
height = rect.get_height()
#ax.annotate('{}'.format(height), #antigo
ax.annotate('{:.0f}'.format(height), #Modificamos para apresentar o número inteiro
xy=(rect.get_x() + rect.get_width() / 2, height),
xytext=(0, 3), # 3 points vertical offset
textcoords="offset points",
ha='center', va='bottom')
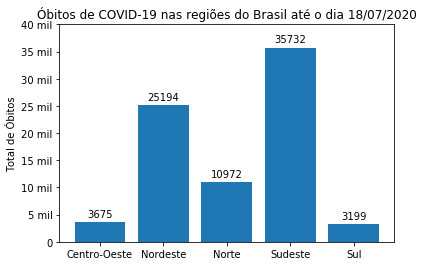
covid_Regioes = covid_BR[['regiao','obitosNovos']].groupby('regiao').sum().query('regiao != "Brasil"')/2
fig, ax = plt.subplots()
plt.ylim(0, 40000) # aumentamos o limite da coordenada y
retangulos = ax.bar(covid_Regioes.index, covid_Regioes['obitosNovos'])
ax.yaxis.set_major_formatter(FuncFormatter(inserir_mil))
ax.set_ylabel('Total de Óbitos') # Rótulo do eixo y
ax.set_title("Óbitos de COVID-19 nas regiões do Brasil até o dia 18/07/2020")
autolabel(retangulos)
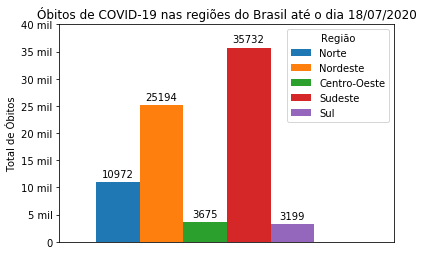
Para realizarmos os “plots” agrupados das barras devemos realizar 5 “plots” distintos, um para cada barra.
Cada plot sofrerá uma translação (exceto o do meio).
Iremos reduzir a largura de cada barra.
covid_Regioes = covid_BR[['regiao','obitosNovos']].groupby('regiao').sum().query('regiao != "Brasil"')/2
largura = 0.3
fig, ax = plt.subplots()
retangulo1 = ax.bar([-2*largura], covid_Regioes.loc[['Norte'],['obitosNovos']].to_numpy()[0], largura, label='Norte')
retangulo2 = ax.bar([-largura], covid_Regioes.loc[['Nordeste'],['obitosNovos']].to_numpy()[0], largura, label='Nordeste')
retangulo3 = ax.bar([0], covid_Regioes.loc[['Centro-Oeste'],['obitosNovos']].to_numpy()[0], largura, label='Centro-Oeste')
retangulo4 = ax.bar([largura], covid_Regioes.loc[['Sudeste'],['obitosNovos']].to_numpy()[0], largura, label='Sudeste')
retangulo5 = ax.bar([2*largura], covid_Regioes.loc[['Sul'],['obitosNovos']].to_numpy()[0], largura, label='Sul')
ax.yaxis.set_major_formatter(FuncFormatter(inserir_mil))
ax.set_ylabel('Total de Óbitos') # Rótulo do eixo y
ax.set_title("Óbitos de COVID-19 nas regiões do Brasil até o dia 18/07/2020")
autolabel(retangulo1); autolabel(retangulo2); autolabel(retangulo3); autolabel(retangulo4); autolabel(retangulo5)
plt.ylim(0, 40000) # aumentamos o limite da coordenada y
plt.xlim(-1, 1.3) # Limites que iremos utilizar na coordenada y
plt.xticks([], []) # Remover os "ticks" no eixo x
#plt.xticks([0], ['Região']) # Se quisermos incluir o rótulo "Região" na posição 0 do eixo x
ax.legend(title="Região")
<matplotlib.legend.Legend at 0x7fa283b23250>
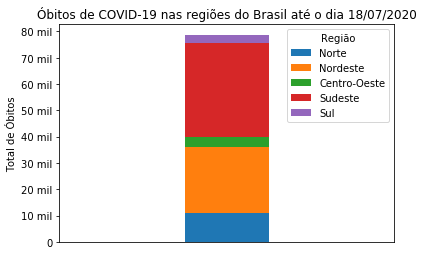
Para empilharmos as barras manualmente devemos utilizar o argumento bottom:
largura = 0.25
obitos_norte = covid_Regioes.loc[['Norte'],['obitosNovos']].to_numpy()[0]
obitos_nordeste = covid_Regioes.loc[['Nordeste'],['obitosNovos']].to_numpy()[0]
obitos_centro_oeste = covid_Regioes.loc[['Centro-Oeste'],['obitosNovos']].to_numpy()[0]
obitos_sudeste = covid_Regioes.loc[['Sudeste'],['obitosNovos']].to_numpy()[0]
obitos_sul = covid_Regioes.loc[['Sul'],['obitosNovos']].to_numpy()[0]
fig, ax = plt.subplots()
retangulo1 = ax.bar([0.5], obitos_norte, largura, label='Norte')
retangulo2 = ax.bar([0.5], obitos_nordeste, largura, label='Nordeste', bottom = obitos_norte)
retangulo3 = ax.bar([0.5], obitos_centro_oeste, largura, label='Centro-Oeste', bottom = obitos_norte + obitos_nordeste)
retangulo4 = ax.bar([0.5], obitos_sudeste, largura, label='Sudeste', bottom = obitos_norte +
obitos_nordeste + obitos_centro_oeste)
retangulo5 = ax.bar([0.5], obitos_sul, largura, label='Sul', bottom = obitos_norte +
obitos_nordeste + obitos_centro_oeste + obitos_sudeste)
ax.yaxis.set_major_formatter(FuncFormatter(inserir_mil))
ax.set_ylabel('Total de Óbitos') # Rótulo do eixo y
ax.set_title("Óbitos de COVID-19 nas regiões do Brasil até o dia 18/07/2020")
plt.xticks([], [])
#plt.xticks([0], ['Região']) # Se quisermos incluir o rótulo "Região" na posição 0 do eixo x
plt.xlim(0,1)
ax.legend(title="Região")
<matplotlib.legend.Legend at 0x7fa28c036b50>
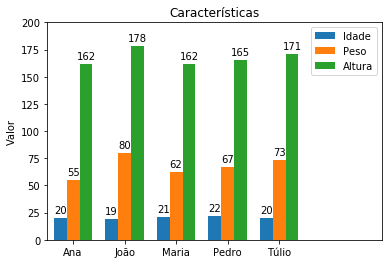
x = np.arange(len(df_dict_series.index))
largura = 0.25
fig, ax = plt.subplots()
retangulo1 = ax.bar(x - largura, df_dict_series.Idade, largura, label='Idade')
retangulo2 = ax.bar(x, df_dict_series.Peso, largura, label='Peso')
retangulo3 = ax.bar(x + largura, df_dict_series.Altura, largura, label='Altura')
autolabel(retangulo1); autolabel(retangulo2); autolabel(retangulo3)
plt.ylim(0,200)
plt.xlim(-0.5,6)
ax.set_ylabel('Valor')
ax.set_title('Características')
ax.set_xticks(x)
ax.set_xticklabels(df_dict_series.index)
ax.legend()
<matplotlib.legend.Legend at 0x7fa28801bad0>
x = np.arange(len(df_dict_series.index))
largura = 0.25
fig, ax = plt.subplots()
retangulo1 = ax.bar(x, df_dict_series.Idade, largura, label='Idade')
retangulo2 = ax.bar(x, df_dict_series.Peso, largura, label='Peso', bottom = df_dict_series.Idade)
retangulo3 = ax.bar(x, df_dict_series.Altura, largura, label='Altura', bottom = df_dict_series.Idade + df_dict_series.Peso)
plt.xlim(-0.5,6)
ax.set_ylabel('Valor')
ax.set_title('Características')
ax.set_xticks(x)
ax.set_xticklabels(df_dict_series.index)
ax.legend()
<matplotlib.legend.Legend at 0x7fa28117e090>
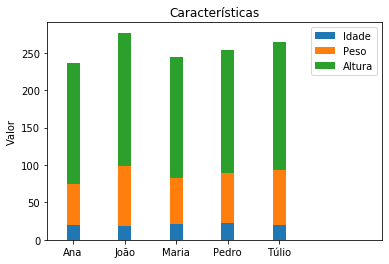
Para construir os gráficos de barras procedemos de maneira análoga ao que foi feito acima.
Substituímos o método bar por barh
Caso haja interesse deve modificar a função autolabel, alterando a altura, height, pela largura, width.
Gráfico de Setores¶
Neste caso devemos modificar o DataFrame para conter percentuais (ou pesos).
Vamos usar os parâmetros:
autopct que adiciona o percentual de cada “fatia”.
shadow que adiciona sombra
explode que separa fatias selecionadas
df_dict_series_pct = df_dict_series.copy()
df_dict_series_pct.Idade = df_dict_series_pct.Idade/df_dict_series_pct.Idade.sum()
df_dict_series_pct.Peso = df_dict_series_pct.Peso/df_dict_series_pct.Peso.sum()
df_dict_series_pct.Altura = df_dict_series_pct.Altura/df_dict_series_pct.Altura.sum()
df_dict_series_pct
| Idade | Peso | Altura | |
|---|---|---|---|
| Ana | 0.196078 | 0.163205 | 0.193317 |
| João | 0.186275 | 0.237389 | 0.212411 |
| Maria | 0.205882 | 0.183976 | 0.193317 |
| Pedro | 0.215686 | 0.198813 | 0.196897 |
| Túlio | 0.196078 | 0.216617 | 0.204057 |
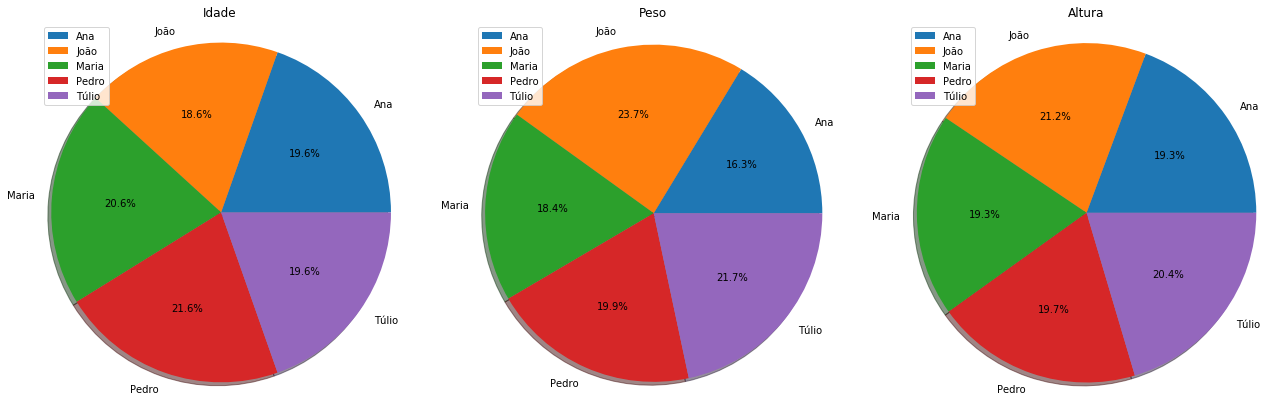
figs, axs = plt.subplots(1,3, figsize=(22,7)) #1 linha3 e 3 colunas de "plots"
axs[0].pie(df_dict_series_pct.Idade, labels=df_dict_series_pct.index, autopct='%1.1f%%', shadow=True)
axs[0].axis('equal') # Igualando os eixos para garantir que obteremos um círculo
axs[0].legend(loc = 'upper left')
axs[0].set_title('Idade')
axs[1].pie(df_dict_series_pct.Peso, labels=df_dict_series_pct.index, autopct='%1.1f%%', shadow=True)
axs[1].axis('equal')
axs[1].legend(loc = 'upper left')
axs[1].set_title('Peso')
axs[2].pie(df_dict_series_pct.Altura, labels=df_dict_series_pct.index, autopct='%1.1f%%', shadow=True)
axs[2].axis('equal')
axs[2].legend(loc = 'upper left')
_ = axs[2].set_title('Altura') #Atribuímos a uma variável para não termos saída
covid_Regioes_pct = covid_Regioes/covid_Regioes.sum()
covid_Regioes_pct['explodir'] = covid_Regioes_pct.index.map(lambda regiao: 0.1 if regiao == 'Nordeste' else 0)
covid_Regioes_pct
| obitosNovos | explodir | |
|---|---|---|
| regiao | ||
| Centro-Oeste | 0.046654 | 0.0 |
| Nordeste | 0.319834 | 0.1 |
| Norte | 0.139288 | 0.0 |
| Sudeste | 0.453613 | 0.0 |
| Sul | 0.040611 | 0.0 |
fig, ax = plt.subplots(figsize = (10,10))
ax.pie(covid_Regioes_pct.obitosNovos, explode=covid_Regioes_pct.explodir,
labels=covid_Regioes_pct.index, autopct='%1.1f%%', shadow=True)
ax.set_title('Percentual de Óbitos de COVID-19 nas Regiões do Brasil até o Dia 18/07/2020')
_ = ax.axis('equal')
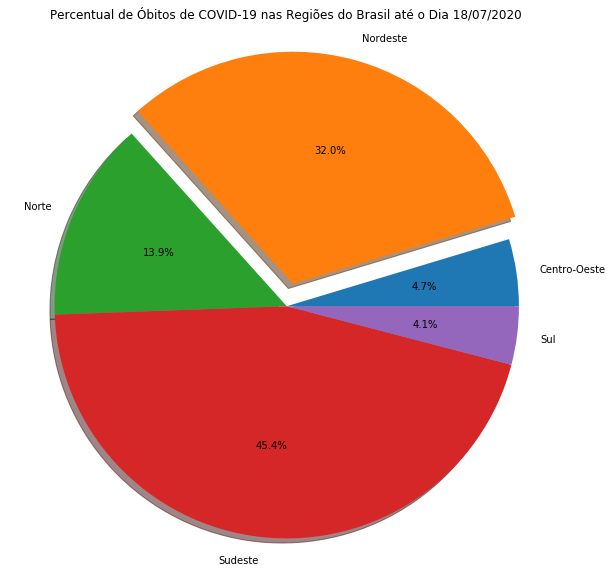
Gráfico de Dispersão¶
Para gráficos de dispersão vários argumentos são os mesmos que já vimos no método plot do pandas.
fig, ax = plt.subplots()
ax.scatter(df_exemplo.index, df_exemplo['coluna_1'])
fig.autofmt_xdate()
ax.set_xlabel('Data')
ax.set_ylabel('Valores da Coluna 1')
ax.set_title('Gráfico do df_exemplo')
Text(0.5, 1.0, 'Gráfico do df_exemplo')
fig, ax = plt.subplots()
ax.scatter(df_exemplo.index, df_exemplo['coluna_1'], s = np.abs(df_exemplo['coluna_2'])*100)
fig.autofmt_xdate()
ax.set_xlabel('Data')
ax.set_ylabel('Valores da Coluna 1')
ax.set_title('Gráfico do df_exemplo')
Text(0.5, 1.0, 'Gráfico do df_exemplo')
covid_PB_casos_obitos = covid_PB[['obitosNovos', 'casosNovos']].sort_index()
fig, ax = plt.subplots()
grafico = ax.scatter(covid_PB_casos_obitos.index, covid_PB_casos_obitos.casosNovos, c = covid_PB_casos_obitos.obitosNovos)
fig.autofmt_xdate()
ax.set_xlabel('Data')
ax.set_ylabel('Casos COVID-19 em PB')
ax.set_title('Casos e Óbitos de COVID-19 na Paraíba')
color_map=ax.get_children()[4]
plt.colorbar(grafico, label = 'Óbitos')
<matplotlib.colorbar.Colorbar at 0x7fa271177550>

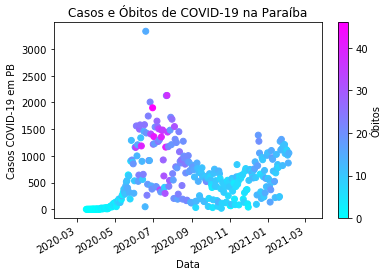
covid_PB_casos_obitos = covid_PB[['obitosNovos', 'casosNovos']].sort_index()
fig, ax = plt.subplots()
grafico = ax.scatter(covid_PB_casos_obitos.index, covid_PB_casos_obitos.casosNovos, c = covid_PB_casos_obitos.obitosNovos,
cmap='cool')
fig.autofmt_xdate()
ax.set_xlabel('Data')
ax.set_ylabel('Casos COVID-19 em PB')
ax.set_title('Casos e Óbitos de COVID-19 na Paraíba')
color_map=ax.get_children()[4]
plt.colorbar(grafico, label = 'Óbitos')
<matplotlib.colorbar.Colorbar at 0x7fa270830690>
fig, ax = plt.subplots()
ax.scatter(df_exemplo.index, df_exemplo['coluna_1'], label = 'Coluna 1', color = 'black')
ax.scatter(df_exemplo.index, df_exemplo['coluna_2'], label = 'Coluna 2', color = 'red')
ax.scatter(df_exemplo.index, df_exemplo['coluna_3'], label = 'Coluna 3', color = 'green')
fig.autofmt_xdate()
ax.legend()
ax.set_ylabel("Valor")
ax.set_xlabel("Data")
Text(0.5, 0, 'Data')
Gráficos Lado a Lado¶
#Vamos modificar esta função para podermos utilizá-la quando temos mais de um gráfico ao mesmo tempo
def autolabel(rects, ax):
"""Attach a text label above each bar in *rects*, displaying its height."""
for rect in rects:
height = rect.get_height()
#ax.annotate('{}'.format(height), #antigo
ax.annotate('{:.0f}'.format(height), #Modificamos para apresentar o número inteiro
xy=(rect.get_x() + rect.get_width() / 2, height),
xytext=(0, 3), # 3 points vertical offset
textcoords="offset points",
ha='center', va='bottom')
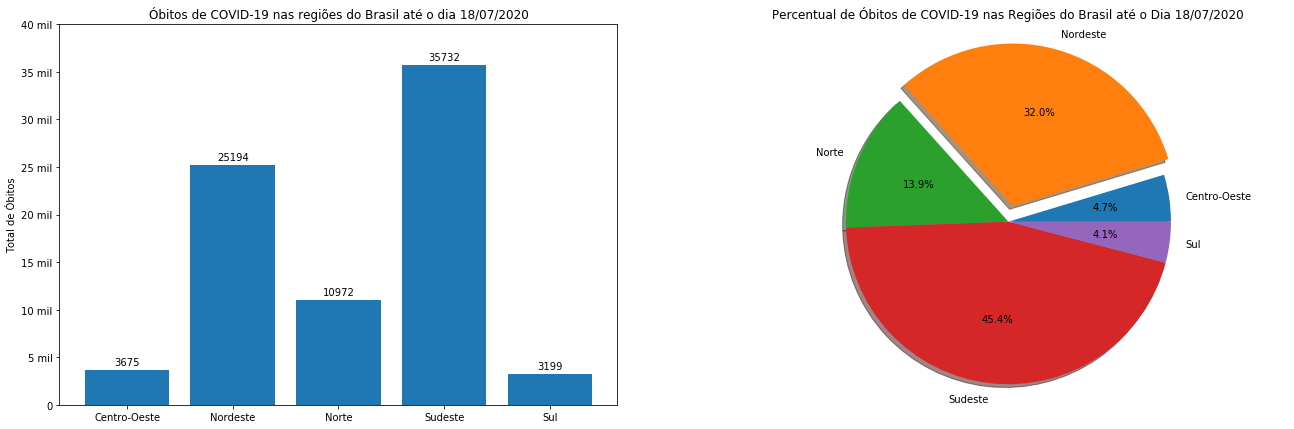
covid_Regioes = covid_BR[['regiao','obitosNovos']].groupby('regiao').sum().query('regiao != "Brasil"')/2
figs, axs = plt.subplots(1,2, figsize=(22,7))
axs[0].set_ylim(0, 40000) # aumentamos o limite da coordenada y
retangulos = axs[0].bar(covid_Regioes.index, covid_Regioes['obitosNovos'])
axs[0].yaxis.set_major_formatter(FuncFormatter(inserir_mil))
axs[0].set_ylabel('Total de Óbitos') # Rótulo do eixo y
axs[0].set_title("Óbitos de COVID-19 nas regiões do Brasil até o dia 18/07/2020")
autolabel(retangulos, axs[0])
axs[1].pie(covid_Regioes_pct.obitosNovos, explode=covid_Regioes_pct.explodir,
labels=covid_Regioes_pct.index, autopct='%1.1f%%', shadow=True)
axs[1].set_title('Percentual de Óbitos de COVID-19 nas Regiões do Brasil até o Dia 18/07/2020')
_ = axs[1].axis('equal')
Histograma¶
fig, ax = plt.subplots()
ax.hist(covid_regioes.obitos_Nordeste, bins=30, color='lime')
ax.set_ylabel('Frequência')
ax.set_xlabel('Óbitos Diários por COVID-19')
ax.set_title('Nordeste')
Text(0.5, 1.0, 'Nordeste')
fig, axs = plt.subplots(1,2, sharey=True, figsize = (15,7)) #sharey=True indica que o eixo y será o mesmo para todos os gráficos
axs[0].hist(covid_regioes.obitos_Nordeste, bins=30, histtype='step', color='red')
axs[0].set_ylabel('Frequência')
axs[0].set_xlabel('Óbitos Diários por COVID-19')
axs[0].set_title('Nordeste')
axs[1].hist(covid_regioes.obitos_Nordeste, bins=30, fill=False, edgecolor='red')
axs[1].set_ylabel('Frequência')
axs[1].set_xlabel('Óbitos Diários por COVID-19')
axs[1].set_title('Nordeste')
Text(0.5, 1.0, 'Nordeste')
fig, ax = plt.subplots(figsize=(10,10))
ax.hist([covid_regioes.obitos_Nordeste, covid_regioes.obitos_Sudeste], bins=30, histtype='step', label=['Nordeste', 'Sudeste'])
ax.set_ylabel('Frequência')
ax.set_xlabel('Óbitos Diários por COVID-19')
_ = ax.legend()
BoxPlot¶
O método para criar o boxplot utilizando o matplotlib se resume a fornecer uma lista (ou similar) de valores para os quais queremos os boxplots e uma lista (ou similar) contendo as posições nas quais queremos que os boxplots apareçam.
fig, ax = plt.subplots()
dados = [df_exemplo['coluna_1'], df_exemplo['coluna_2'], df_exemplo['coluna_3'].dropna()]
posicoes = np.array(range(len(dados))) + 1
ax.boxplot(dados, positions=posicoes)
_ = ax.set_xticklabels(['Coluna 1', 'Coluna 2', 'Coluna 3'])
/Users/gustavo/anaconda3/lib/python3.7/site-packages/numpy/core/_asarray.py:83: VisibleDeprecationWarning: Creating an ndarray from ragged nested sequences (which is a list-or-tuple of lists-or-tuples-or ndarrays with different lengths or shapes) is deprecated. If you meant to do this, you must specify 'dtype=object' when creating the ndarray
return array(a, dtype, copy=False, order=order)
covid_norte = covid_regioes.obitos_Norte
covid_nordeste = covid_regioes.obitos_Nordeste
covid_sudeste = covid_regioes.obitos_Sudeste
covid_sul = covid_regioes.obitos_Sul
covid_centro_oeste = covid_regioes['obitos_Centro-Oeste']
covid_box = [covid_norte, covid_nordeste, covid_sudeste, covid_sul, covid_centro_oeste]
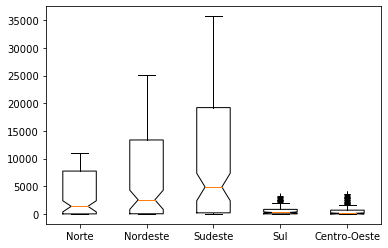
fig, ax = plt.subplots()
posicoes = np.array(range(len(covid_box))) + 1
ax.boxplot(covid_box, 1, positions=posicoes, sym='+')
_ = ax.set_xticklabels(['Norte', 'Nordeste', 'Sudeste', 'Sul', 'Centro-Oeste'])
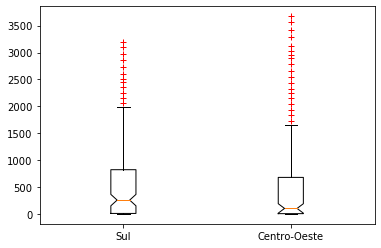
covid_box_2 = [covid_sul, covid_centro_oeste]
fig, ax = plt.subplots()
posicoes = np.array(range(len(covid_box_2))) + 1
ax.boxplot(covid_box_2, 1, positions=posicoes, sym='r+') #r indica 'red', vermelho, + é o símbolo para o outlier
_ = ax.set_xticklabels(['Sul', 'Centro-Oeste'])
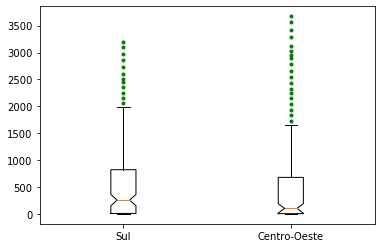
covid_box_2 = [covid_sul, covid_centro_oeste]
fig, ax = plt.subplots()
posicoes = np.array(range(len(covid_box_2))) + 1
ax.boxplot(covid_box_2, 1, positions=posicoes, sym='g.') #r indica 'red', vermelho, + é o símbolo para o outlier
_ = ax.set_xticklabels(['Sul', 'Centro-Oeste'])
Obs.: Muitos dos argumentos utilizados nos métodos acima também funcionam no método plot do pandas, vale a pena testar!