Criação de Representações Visuais e Estética
Contents
3. Criação de Representações Visuais e Estética#
Neste capítulo, consideraremos o modelo básico para representação visual, rudimentos das representações visuais em uma, duas e três dimensões, tipos de dados e estética.
3.1. Modelo referencial#
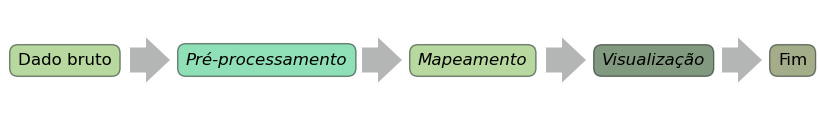
A geração de representações visuais através do computador segue um modelo referencial geralmente descrito pelas seguintes componentes:
Entrada: dado bruto à disposição do storyteller que será manipulado para produzir um visual.
Pré-Processamento: etapa secundária de preparação e tratamento dos dados.
Mapeamento: etapa terciária onde são definidos o projeto gráfico, as estruturas visuais e o mapeamento de valores em formas quantificadas que determinarão a estética.
Visualização: etapa finalística que entrega o produto final, isto é, a própria representação visual.
A figura abaixo – criada através de objetos patch do matplotlib – ilustra o fluxograma do modelo referencial. Como podemos ver pelo código acompanhante, a representação visual é totalmente gerada via Python e não passou por nenhum outro software de desenho. Este exemplo é uma constatação de quão poderoso o matplotlib é. Porém, criar figuras de maneira elementar exige tempo e dedicação. Nem sempre será necessário seguir este caminho para criarmos bons gráficos, mas aplicar-se a conhecer a ferramenta em profundidade enaltece a capacidade técnica de engenharia visual.
Detalharemos as etapas importantes nas subseções a seguir.
import matplotlib.colors as clr
import matplotlib.pyplot as plt
import matplotlib.patches as pch
from matplotlib.path import Path
def draw_my_arrow(A1:tuple, B1:tuple, C1:tuple, D:tuple, C2:tuple, B2:tuple, A2:tuple):
"""
Desenha uma seta com 7 vértices com largura e comprimento controláveis.
C1
o .
A1 B1| .
o - - - - o .
| o D
o - - - - o .
A2 B2| .
o .
C2
Larg. corpo: m(A1A2)
Compr. corpo: m(A1B1)
Larg. cabeça: m(B1C1)
Compr. cabeça: m(B1D)
"""
# pontos
points = [A1, B1, C1, D, C2, B2, A2]
# caminho
moves = [Path.MOVETO, Path.LINETO, Path.LINETO, Path.LINETO, Path.LINETO, Path.LINETO, Path.LINETO]
return Path(points, moves)
# mapa de cores XKCD
xkcd = clr.XKCD_COLORS
# parâmetros
x0,y0 = 0,0 # origem
xa,ya = 1, 1 # 100% da área da figura
L,H = 8, 1 # tamanho da figura (polegadas)
# propriedades de my_arrow
xmid, ymid = 0.15, 0.5
body_width, body_length = 0.25, 0.02
head_width, head_length = 0.1, 0.03
# figura e eixo
fig = plt.figure(figsize=(L,H))
ax = fig.add_axes(rect=[x0, y0, xa, ya], frameon=False)
ax.set_axis_off()
'''CRIAÇÃO DE ELEMENTOS VISUAIS'''
# DADO BRUTO
ax.text(0.01,0.45,
s='Dado bruto',
size=12,
style='normal',
bbox={'facecolor': xkcd['xkcd:nasty green'],
'edgecolor':xkcd['xkcd:almost black'],
'alpha': 0.5,
'boxstyle': 'round',
'pad': 0.5})
# SETA 1
arrow = draw_my_arrow(A1=(xmid,ymid + body_width/2),
B1=(xmid + body_length, ymid + body_width/2),
C1=(xmid + body_length, ymid + body_width/2 + head_width),
D=(xmid + body_length + head_length, ymid),
C2=(xmid + body_length, ymid - body_width/2 - head_width),
B2=(xmid + body_length, ymid - body_width/2),
A2=(xmid,ymid - body_width/2)
)
arrow = pch.PathPatch(arrow,facecolor=xkcd['xkcd:almost black'],lw=0,alpha=0.3)
ax.add_patch(arrow)
# PRÉ-PROCESSAMENTO
ax.text(0.22,0.45,
s='Pré-processamento',
style='italic',
size=12,
bbox={'facecolor': xkcd['xkcd:algae green'],
'edgecolor':xkcd['xkcd:almost black'],
'alpha': 0.5,
'boxstyle': 'round',
'pad': 0.5})
# SETA 2
xmid += 0.29
arrow = draw_my_arrow(A1=(xmid,ymid + body_width/2),
B1=(xmid + body_length, ymid + body_width/2),
C1=(xmid + body_length, ymid + body_width/2 + head_width),
D=(xmid + body_length + head_length, ymid),
C2=(xmid + body_length, ymid - body_width/2 - head_width),
B2=(xmid + body_length, ymid - body_width/2),
A2=(xmid,ymid - body_width/2)
)
arrow = pch.PathPatch(arrow,facecolor=xkcd['xkcd:almost black'],lw=0,alpha=0.3)
ax.add_patch(arrow)
# MAPEAMENTO
ax.text(0.51,0.45,
s='Mapeamento',
style='italic',
size=12,
bbox={'facecolor': xkcd['xkcd:nasty green'],
'edgecolor':xkcd['xkcd:almost black'],
'alpha': 0.5,
'boxstyle': 'round',
'pad': 0.5})
# SETA 3
xmid += 0.23
arrow = draw_my_arrow(A1=(xmid,ymid + body_width/2),
B1=(xmid + body_length, ymid + body_width/2),
C1=(xmid + body_length, ymid + body_width/2 + head_width),
D=(xmid + body_length + head_length, ymid),
C2=(xmid + body_length, ymid - body_width/2 - head_width),
B2=(xmid + body_length, ymid - body_width/2),
A2=(xmid,ymid - body_width/2)
)
arrow = pch.PathPatch(arrow,facecolor=xkcd['xkcd:almost black'],lw=0,alpha=0.3)
ax.add_patch(arrow)
# VISUALIZAÇÃO
ax.text(0.74,0.45,
s='Visualização',
style='italic',
size=12,
bbox={'facecolor': xkcd['xkcd:dark green'],
'edgecolor':xkcd['xkcd:almost black'],
'alpha': 0.5,
'boxstyle': 'round',
'pad': 0.5})
# SETA 4
xmid += 0.22
arrow = draw_my_arrow(A1=(xmid,ymid + body_width/2),
B1=(xmid + body_length, ymid + body_width/2),
C1=(xmid + body_length, ymid + body_width/2 + head_width),
D=(xmid + body_length + head_length, ymid),
C2=(xmid + body_length, ymid - body_width/2 - head_width),
B2=(xmid + body_length, ymid - body_width/2),
A2=(xmid,ymid - body_width/2)
)
arrow = pch.PathPatch(arrow,facecolor=xkcd['xkcd:almost black'],lw=0,alpha=0.3)
ax.add_patch(arrow)
# FIM
ax.text(0.96,0.45,
s='Fim',
size=12,
style='normal',
bbox={'facecolor': xkcd['xkcd:army green'],
'edgecolor':xkcd['xkcd:almost black'],
'alpha': 0.5,
'boxstyle': 'round',
'pad': 0.5});
3.1.1. Dado bruto#
Empregamos o termo “dado bruto” (ou “dado cru”) para fazer referência a toda sorte de dado disponível no mundo que não passou por nenhum processo de tratamento ou limpeza. Dados brutos raramente possuem uma estrutura lógica que facilita a compreensão de como estão organizados. Quando minimamente organizados, a melhor forma de manipulá-los é através de tabelas, serializações, ou outro formato apropriado capaz de gerar datasets.
3.1.1.1. Exemplo 1: registros cirúrgicos e obstétricos de um hospital#
O arquivo raw-data-hospital.txt a seguir mostra um exemplo de dado bruto. Nele foram registrados os números de cirurgias (C) e partos (P) realizados nos blocos 1 (BLC1), 2 (BLC2) e 3 (BLC3) de um hospital público de grande porte no primeiro semestre de 2021 que levaram até 2 horas (I) ou mais do que 2 horas (II) de intervenção.
-- raw-data-hospital.txt
:BLC1 01/Fev/2021 I 30C 2P
:BLC2 20/Fev/2021 II 0C 3P
:BLC1 02/Mar/2021 I 0C 1P
:BLC3 03/Mar/2021 II 0C 0P
:BLC1 22/Abr/2021 I 7C 2P
:BLC1 21-Abr/2021 II 1C 0p
:BLC2 12-Mai/2021 I 22C 10P
:BLC3 22-Mai/2021 II 1C 5P
:BLC3 11/06/2021 I 36C 1P
:blc2 01/06/2021 II 11C 22P
.blc1 12.Jun.2021 I 33c 20P
.blc2 30.Jun.2021 II 0C 17P
.blc1 15.Jun.2021 I 42c 32P
Como podemos ver pelo conteúdo, é difícil ter uma visualização clara das informações nele contidas. Além disso, ele possui uma diversidade de inconsistências de caracteres, produzindo uma formatação não padronizada. Antes de o utilizarmos para propósitos de visualização, devemos realizar operações de tratamento.
3.1.1.2. Exemplo 2: monitoramento climático em tempo real#
Há ocasiões em que arquivos bem estruturados estão disponíveis, mas, mesmo assim, é preciso algum tipo de manipulação para extrair deles a visualização desejada. Abaixo temos um extrato do arquivo NEONDSTowerTemperatureData.hdf5, que possui registros de temperatura coletados por sensores localizados em torres de captação (Fig. 3.1) de dois domínios ecoclimáticos terrestres dos EUA (03: Flórida e 10: Colorado).
O arquivo pleno pode ser baixado do site da National Ecolological Observatory Network. Trata-se de um dado formatado hierarquicamente, com extensão hdf (Hierarchical Data Format). Arquivos HDF podem ser entendidos como um grande diretório compactado que pode conter uma árvore de subdiretórios em muitos níveis. Eles possuem datasets contendo objetos e atributos que podem ser agrupados e linkados internamente de forma variada. Neste curso, não faremos uma análise profunda sobre este formato, mas daremos exemplos de uso.

Fig. 3.1 Torre de monitoramento climático com sensores (boom) distribuídos em 4 diferentes alturas e o quinto sensor posicionado no topo da torre, para captação de temperatura dos domínios ecoclimáticos terrestres americanos. Fonte: National Ecological Observatory Network/EUA.#
# importa módulo para ler HDF
import h5py as h5
# lê arquivo
file_hdf = h5.File('../data/NEONDSTowerTemperatureData.hdf5','r')
# imprime valores na hierarquia do domínio '03: Flórida'
for i in range(5):
print(file_hdf['Domain_03']['OSBS']['min_1']['boom_1']['temperature'][i])
(b'2014-04-01 00:00:00.0', 60, 15.061538467566288, 14.968860863452065, 15.156246969609782, 0.00265501529680849, 0.006652086510773004, 0.01620325065082839)
(b'2014-04-01 00:01:00.0', 60, 14.998577866382611, 14.937196648312714, 15.042742647434968, 0.0012541174110136926, 0.0045718657952993555, 0.013061110131184767)
(b'2014-04-01 00:02:00.0', 60, 15.262312876008354, 15.035021092564822, 15.566825685692056, 0.041437536746907304, 0.026279756704514122, 0.05349681721402945)
(b'2014-04-01 00:03:00.0', 60, 15.453513600198388, 15.385534796704409, 15.534492124029654, 0.0011747586052929563, 0.004424851419902813, 0.012868326141695046)
(b'2014-04-01 00:04:00.0', 60, 15.353062311043297, 15.237990731687008, 15.423457606288034, 0.003526442848776773, 0.00766642338249153, 0.01788372021053885)
A tupla armazena, nesta ordem: data e hora da medição, número de pontos de cálculo das estatísticas (60 segundos), temperaturas média, mínima e máxima (por minuto), variância, erro e incerteza. Mesmo assim, para produzirmos alguma visualização útil, temos que perscrutar o arquivo e entender cada um de seus atributos.
3.1.1.2.1. Introspectando arquivos HDF#
A visualização da “árvore” de um arquivo HDF é possível via interface ou linha de comando. No primeiro caso, basta fazer o download da ferramenta HDF View e seguir o guia do usuário. No segundo caso, há pelo menos dois modos práticos:
Modo 1: usando uma função definida pelo usuário para impressão dos objetos e atributos:
# leitura do arquivo
file_hdf = h5.File('../data/NEONDSTowerTemperatureData.hdf5','r')
# função para imprimir conteúdo do arquivo 'file_hdf'
def print_h5_tree(name, obj):
print(name, dict(obj.attrs))
# chamada
file_hdf.visititems(print_h5_tree)
Modo 2: usando o pacote
nexusformat– instalável viapip install nexusformat—, que lida com o formato NeXus:
# importação da função `nxload`
from nexusformat.nexus import nxload
# leitura do arquivo
hdf5 = nxload('../data/NEONDSTowerTemperatureData.hdf5')
# impressão
print(hdf5.tree)
3.1.2. Pré-Processamento#
Como já é sabido de cursos prévios sobre análise de dados, podemos realizar o workflow tradicional com pandas e outros módulos necessários para extração, limpeza, tratamento e carregamento dos dados, a fim de torná-los adequados para manuseio. Podemos aplicar operações de filtragem, casting, splitting de strings, renomeação de atributos, salvamento em formatos tabelares etc.
Exercitando técnicas que já constam em nosso portifólio de conhecimento, podemos trabalhar sobre os arquivos dos exemplos anteriores para obter versões mais claras.
3.1.2.1. Exemplo 1: reorganizando o arquivo de registros cirúrgicos e obstétricos#
import pandas as pd
# leitura de arquivo
df_h = pd.read_csv('../data/raw-data-hospital.txt',sep=' ',names=['Bloco','Data','Porte','Cirurgias','Partos'])
# remoção de inconsistências nas séries
df_h['Bloco'] = df_h['Bloco'].str.upper().str.strip(':BLC').str.strip('.BLC')
df_h['Data'] = df_h['Data'].str.replace('-','/').str.replace('.','/').\
str.replace('/','-').str.replace('06','Jun').\
str.replace(r'([A-Z][a-z][a-z])',lambda x: x.group().lower(),regex=True)
df_h['Cirurgias'] = df_h['Cirurgias'].str.strip('C').str.strip('c')
df_h['Partos'] = df_h['Partos'].str.strip('P').str.strip('p')
# reindexação
df_h.set_index('Data',inplace=True)
A tabela que resulta do processamento realizado pelo código acima é a seguinte:
| Bloco | Porte | Cirurgias | Partos | |
|---|---|---|---|---|
| Data | ||||
| 01-fev-2021 | 1 | I | 30 | 2 |
| 20-fev-2021 | 2 | II | 0 | 3 |
| 02-mar-2021 | 1 | I | 0 | 1 |
| 03-mar-2021 | 3 | II | 0 | 0 |
| 22-abr-2021 | 1 | I | 7 | 2 |
| 21-abr-2021 | 1 | II | 1 | 0 |
| 12-mai-2021 | 2 | I | 22 | 10 |
| 22-mai-2021 | 3 | II | 1 | 5 |
| 11-jun-2021 | 3 | I | 36 | 1 |
| 01-jun-2021 | 2 | II | 11 | 22 |
| 12-jun-2021 | 1 | I | 33 | 20 |
| 30-jun-2021 | 2 | II | 0 | 17 |
| 15-jun-2021 | 1 | I | 42 | 32 |
3.1.2.2. Exemplo 2: acessando dados de temperatura#
Como o arquivo HDF possui uma hierarquia bem definida, podemos usar keys para acessar os datasets e usar métodos do numpy e do pandas para recuperar a estrutura e criar os dataframes diretamente.
from pandas import DataFrame
from numpy import array
# conversão para array e posterior criação do dataframe
df_t = DataFrame(array(file_hdf['Domain_10']['STER']['min_1']['boom_2']['temperature']))
df_t.set_index('date')
| numPts | mean | min | max | variance | stdErr | uncertainty | |
|---|---|---|---|---|---|---|---|
| date | |||||||
| b'2014-04-01 00:00:00.0' | 60 | 6.398707 | 6.347253 | 6.440027 | 0.001093 | 0.004269 | 0.012512 |
| b'2014-04-01 00:01:00.0' | 60 | 6.375695 | 6.305179 | 6.439385 | 0.002014 | 0.005793 | 0.014728 |
| b'2014-04-01 00:02:00.0' | 60 | 6.360941 | 6.293467 | 6.430070 | 0.002428 | 0.006361 | 0.015624 |
| b'2014-04-01 00:03:00.0' | 60 | 6.384913 | 6.336319 | 6.418260 | 0.000289 | 0.002193 | 0.010185 |
| b'2014-04-01 00:04:00.0' | 60 | 6.283479 | 6.236615 | 6.332181 | 0.000531 | 0.002975 | 0.010937 |
| ... | ... | ... | ... | ... | ... | ... | ... |
| b'2014-04-03 23:56:00.0' | 60 | 6.495163 | 6.486047 | 6.513575 | 0.000053 | 0.000941 | 0.009399 |
| b'2014-04-03 23:57:00.0' | 60 | 6.475617 | 6.460469 | 6.497839 | 0.000119 | 0.001411 | 0.009627 |
| b'2014-04-03 23:58:00.0' | 60 | 6.464438 | 6.456761 | 6.471775 | 0.000020 | 0.000573 | 0.009280 |
| b'2014-04-03 23:59:00.0' | 60 | 6.483857 | 6.464980 | 6.511195 | 0.000177 | 0.001716 | 0.009820 |
| b'2014-04-04 00:00:00.0' | 60 | 6.534577 | 6.512014 | 6.543662 | 0.000088 | 0.001208 | 0.009519 |
4323 rows × 7 columns
3.1.3. Mapeamento#
O mapeamento visual é definido pelo processo de transferir os dados para a área de visualização e exibi-los através de estruturas visuais adequadas. Há muitos dados abstratos, cuja natureza não se vincula a uma estrutura topológica (forma) ou a uma localização geográfica. Nesses casos, a estrutura visual mais adequada pode não ser imediata.
Por exemplo, as temperaturas coletadas do monitoramento ambiental são diretamente associadas com uma localização geográfica dentro do território americano. Os dados de registro hospitalar, embora também possam ser associados a um local físico (hospital), o local é desconhecido. Casos como este não possuem correspondência imediaata com dimensões. Outro exemplo de dado abstrato é, por exemplo, o consumo quilômetro/litro de automóveis que rodam a gasolina.
Há três estruturas fundamentais que consolidam o mapeamento visual:
substrato espacial: define as dimensões do espaço físico onde a representação visual é criada (eixos Cartesianos, por exemplo);
elementos gráficos: caracterizam os elementos visuais que aparecem no espaço;
propriedades gráficas: definem-se pelas propriedades dos elementos gráficos às quais o olho humano (retina) são sensíveis. São também conhecidos como atributos visuais.
3.1.3.1. Categorias e tipos de dados#
Na Seção 1.6.2, elencamos algumas definições para tipos de dados. Neste ponto, vale acomodar o espectro das definições para considerar duas categorias principais e algumas subcategorias da seguinte forma:
Dado quantitativo
Numérico contínuo: valores numéricos (inteiros, racionais, reais);
Numérico discreto: comumemente inteiros, mas podem ser fracionários quando não há valores intervalares a considerar;
Dado qualitativo
Categórico não ordenado: discretos e sem ordem natural. Também chamado de fatores.
Categório ordenado (ordinal): discretos e ordenados. Também chamado de fatores ordenados.
Data, hora e texto são casos especiais que não categorizaremos por, às vezes, se comportarem como quantitativos, qualitativos ou nenhuma das duas categorias.
Alguns exemplos são vistos na Tabela 3.1.
Categoria |
Tipo |
Exemplo |
Descrição |
|---|---|---|---|
Quantitativo |
Numérico contínuo |
2.7,1.22,10,1e-5 |
Valores numéricos arbitrários. |
Quantitativo |
Numérico discreto |
1,2,3; 0.5,1.5, 2.5 |
Números em unidades discretas. Fracionários em escalas especiais. |
Qualitativo |
Categórico não ordenado |
mamífero, crustáceo, aracnídeo |
Categoria únicas, discretas e sem ordem aparente. |
Qualitativo |
Categórico ordenado |
Péssimo, razoável, excelente |
Categoria únicas, discretas e com ordenamento por significado. “Razoável” é algo entre “péssimo” e “excelente”. |
- |
Numérico contínuo/Discreto |
23/03/1996, 13:22 |
Podem ser “contínuos” se considerados em intervalos contíguos (datas sucessivas), ou discretos, se tomados particularmente. |
- |
- |
Tempos vividos não retornam; sim/talvez/não; jamais/sempre. |
Texto em formato livre não possui categoria definida. Em outras situações, pode ser considerado categórico, discreto. |
3.1.3.2. Atributos e elementos visuais#
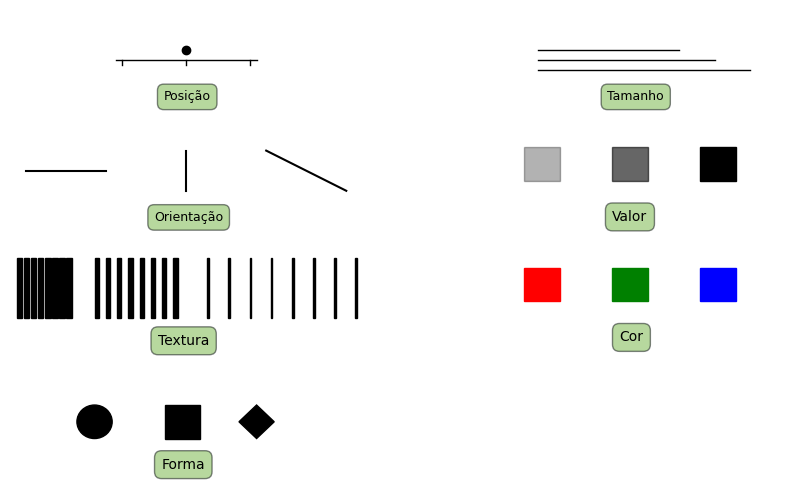
Jacques Bertin estabeleceu os fundamentos teóricos para a visualização da informação elencando 7 atributos visuais, ou canais, (Tabela 3.2):
posição;
tamanho;
orientação;
valor;
textura (ou granularidade);
cor; e
forma.
A figura abaixo, produzida unicamente com matplotlib, ilustra esses conceitos.
Atributo |
Designações |
|---|---|
Posição |
esquerda, centro, direita etc. |
Tamanho |
pequeno, médio, grande etc. |
Orientação |
horizontal, vertical, oblíqua |
Valor |
claro, médio, escuro (tons de cinza) |
Textura |
fina, média, grossa |
Cor |
vermelho, verde, azul etc. |
Forma |
círculo, retângulo, polígono de n lados etc. |
Os elementos visuais possíveis são 4:
ponto;
linha;
superfície; e
volume.
Exemplos desses elementos visuais também puramente criados com matplotlib são desenhados a seguir.

3.1.4. Visualização#
A representação visual é o produto final obtido quando se segue o modelo referencial apresentado anteriormente. Para atingirmos uma visualização eficaz, além de levar em conta todos os aspectos já discutidos na etapa do mapeamento, devemos nos atentar para o bom design e para o propósito da visualização que desenvolveremos ([Mazza, 2009]).
Duas características cruciais a observar em um projeto de visualização são:
reproduzir a informação com fidelidade;
facilitar a compreensão do espectador;
analisar o tipo e a natureza dos dados que serão representados; e
entender a audiência a quem os dados serão direcionados.
Elaborando-se um plano preliminar para a representação visual almejada, a probabilidade de sucesso do projeto é maior. Este plano deve considerar as seguintes variáveis e questões (Tabela 3.3,adaptada deste site):
Definição do problema: que representação é necessária? O que desejo comunicar?
Exame da natureza dos dados: os dados são quantitativos ou qualitativos? Como mapeá-los?
Dimensões: qual é o número de dimensões dos dados (atributos)? Quais são independentes e quais são dependentes? Os dados são univariados (uma dimensão varia com respeito a outra)? Bivariados (uma dimensão varia com respeito a outras duas)? Trivariados (uma dimensão varia com respeito a outras três)? Multivariados (uma dimensão varia com respeito a quatro ou mais dimensões independentes)?
Estrutura de dados: os dados são lineares (codificados em vetores, tabelas, matrizes, coleções etc.)? São temporais (mudam com o tempo)? São geográficos ou espaciais (possuem correspondência com algo físico, como mapas, plantas, projeções de terreno etc.)? São hierárquicos (sugerem organizações, diretórios, genealogias etc.)? Ou são em forma de redes (descrevem relacionamento entre entidades)?
Tipo de interação: a visualização é estática (imagem impressa fisicamente ou em tela de computadores)? Transformável (usuário altera parâmetros que modificam e transformam os dados)? Ou é manipulável (usuário tem possibilidade de girar elementos 3D, aplicar zoom)?
Problema |
Tipo de dado |
Dimensões |
Estrutura de dados |
Tipo de interação |
|---|---|---|---|---|
comunicar |
quantitativo |
univariado |
linear |
estático |
3.1.4.1. Exemplo 1: visualizando o número de cirurgias e partos por bloco#
import seaborn as sns
# reindexação
df_h2 = df_h.reset_index()
# conversão do tipo de dados
df_h2['Cirurgias'] = df_h2['Cirurgias'].astype(int)
df_h2['Partos'] = df_h2['Partos'].astype(int)
# plotagem principal
fig, ax = plt.subplots(1,2,figsize=(10,2))
ax[0].grid(alpha=0.5)
ax[1].grid(alpha=0.5)
p1 = sns.barplot(x='Bloco', y='Cirurgias', hue='Porte', data=df_h2, errorbar=None, palette='Blues', ax=ax[0])
p2 = sns.barplot(x='Bloco', y='Partos', hue='Porte', data=df_h2, errorbar=None, palette='Blues', ax=ax[1])
# eixos
p1.spines['top'].set_visible(False)
p1.spines['right'].set_visible(False)
p1.spines['bottom'].set_position(('axes', -0.1))
p2.spines['top'].set_visible(False)
p2.spines['right'].set_visible(False)
p2.spines['bottom'].set_position(('axes', -0.1))
# legenda
p1.get_legend().remove()
p2.legend(title='Porte',bbox_to_anchor=(1.2,1.0));
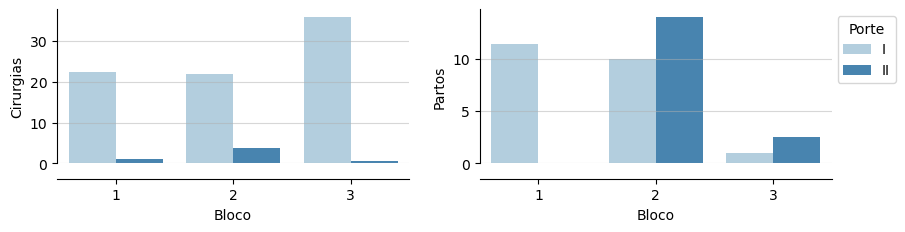
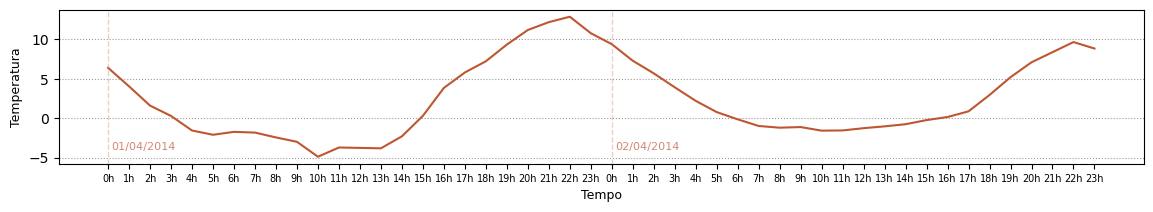
3.1.4.2. Exemplo 2: plotando série temporal de temperatura#
import numpy as np
df_hours = df_t['date'][0::60].index
df_ti = df_t.iloc[df_hours][:48]
fig, ax = plt.subplots(figsize=(14,2))
ax.plot(df_ti['mean'],c='#be5631',lw=1.5)
da = np.array(list((df_ti['mean'].index)))
db = np.array(2*[str(i) + 'h' for i in range(0,24)])
ax.set_xticks(da,db,fontsize=7)
half = int(len(da)/2)
ax.axvline(0,ls='--',lw=1,c='#d1886e',alpha=0.4)
ax.axvline(da[half],ls='--',lw=1,c='#d1886e',alpha=0.4)
ax.grid(axis='y',ls=':',c='k',alpha=0.4)
ax.set_xlabel('Tempo',fontsize=9)
ax.set_ylabel('Temperatura',fontsize=9)
ax.text(10,-4,s='01/04/2014',fontsize=8,c='#d1886e')
ax.text(1450,-4,s='02/04/2014',fontsize=8,c='#d1886e');
3.1.4.3. Exemplo 3: evolução econômica de países#
Este exemplo aborda não uma RV estática, mas transformável e manipulável, ao mesmo tempo.
import plotly.express as px
df = px.data.gapminder()
df.rename(columns={
'country': 'País',
'continent': 'Continente',
'year': 'Ano',
'lifeExp': 'Expectativa de vida',
'pop': 'População',
'gdpPercap': 'PIB per capita'
}, inplace=True)
fig = px.scatter(df, x="PIB per capita", y="Expectativa de vida",
animation_frame="Ano",
animation_group="País",
size="População",
color="Continente",
hover_name="País",
log_x=True,
size_max=55,
range_x=[100,100000],
range_y=[25,90])
fig.update_layout(
template='simple_white',
title="Evolução econômica",
width=650,
height=450,
margin=dict(l=40, r=40, b=40, t=40),
);
3.2. Referências#
- Maz09
Riccardo Mazza. Introduction to information visualization. Springer Science & Business Media, 2009.

